scATAC-seq_v3用户手册
scATAC-seq_v3用户手册
1. 第一章 应用范围
scATAC-seq_v3可以处理以下试剂盒的高通量测序数据分析:
DNBelab C系列高通量单细胞ATAC文库制备试剂盒套装。
2. 第二章 产品介绍
2.1 分析流程图
scATAC-seq_v3分析流程是基于STOmics时空云平台系统开发的一款自动分析流程,其中包括参考基因组比对、反卷积(细胞鉴定与合并)、质控、检峰(peak calling)、下游分析,以及报告生成。
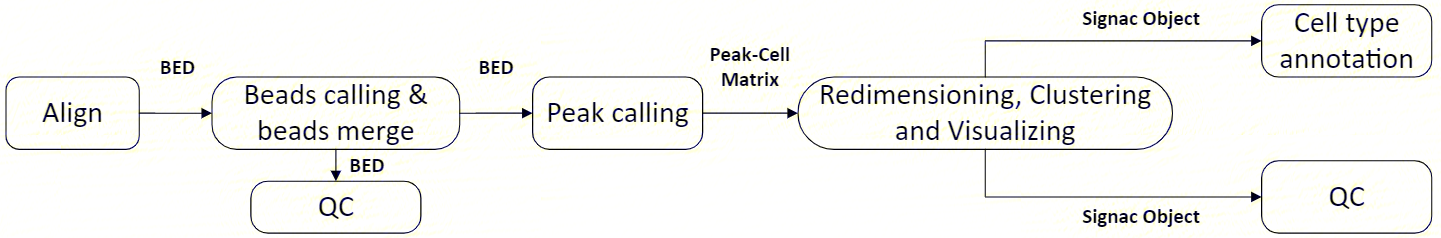
2.2 参考基因组比对
使用chromap (v0.2.3_r407) 将输入的FASTQ文件与参考基因组文件进行比对。
2.2.1 反卷积
使用d2c (v1.4.4) 捕获到完整细胞的beads,再根据beads中fragment中的相似性进行beads合并。
2.2.2 质控
进行片段分布统计和TSS区域富集,进行质控。
2.2.3 检峰
使用 macs2 (v2.2.7.1) 计算方法识别基因组中由测序得到的比对reads富集的区域,对ATAC而言,peak为开放染色质的区域,并生成peak-cell矩阵。
2.2.4 下游分析
基于生成的矩阵进行细胞质控,并进行降维、聚类、细胞类型注释等下游分析。
2.2.5 报告输出
综合分析结果,整理汇总成HTML报告。
3. 第三章 使用说明书
scATAC-seq_v3分析流程通过STOmics时空云平台系统进行样本输入及报告输出的全流程管理。下面具体介绍基于STOmics时空云平台系统使用scATAC-seq_v3分析流程的操作指南。
3.1 指南概述
本章介绍如何使用scATAC-seq_v3分析流程进行分析。在使用之前,请认真阅读并理解其中内容,保证能够正确使用scATAC-seq_v3分析流程。
3.2 使用场景一:手动投递
操作共包括四个步骤:上传数据、构建参考基因组(可选)、样本信息录入、启动分析。完成样本信息录入后,运行任务,当客户在看到任务状态为completed 时,代表任务已完成,即可查看报告部分(详见3.4部分)。
3.2.1 步骤一:上传数据
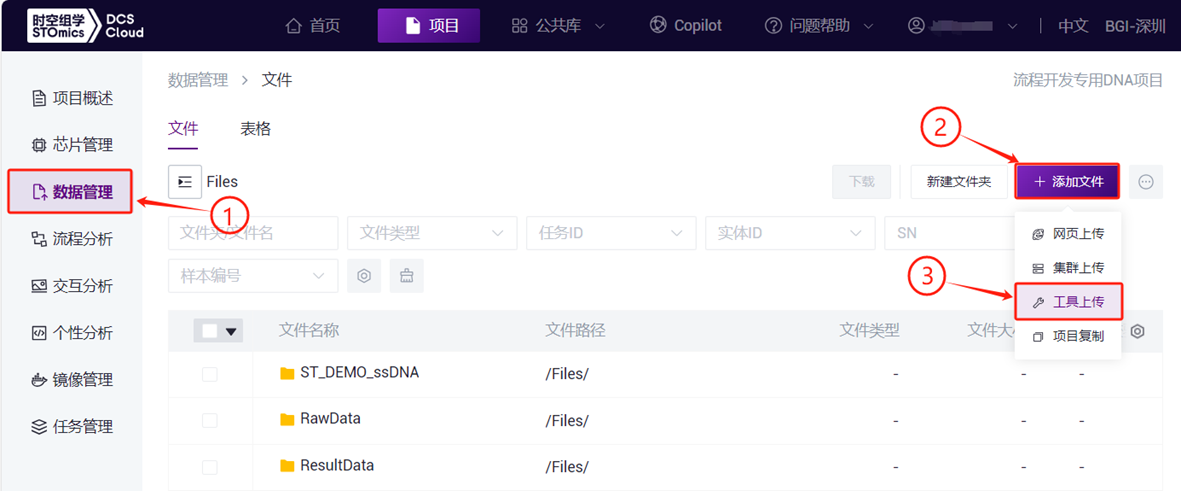
- 点击导航栏**【数据管理】,进入数据管理页面,进入目标文件夹,点击右上角【+添加文件】-【工具上传】**上传数据(图3-1)。
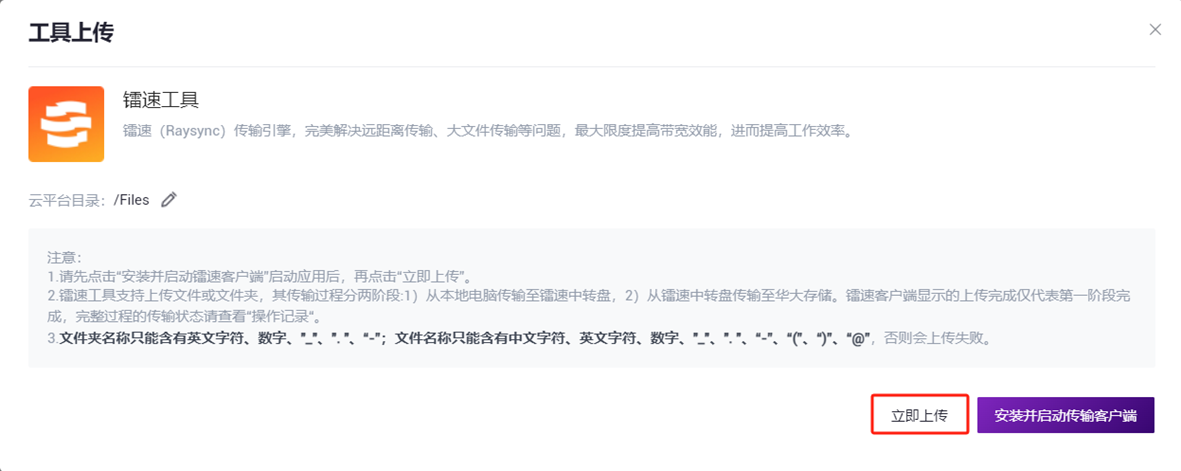
- 点击**【立即上传】浏览并选择所需文件(图3-2),上传完成后文件会在目标文件夹中显示(如首次上传,则需点击【安装并开启传输客户端】**安装所需工具)。
3.2.2 步骤二:构建参考基因组(可选)
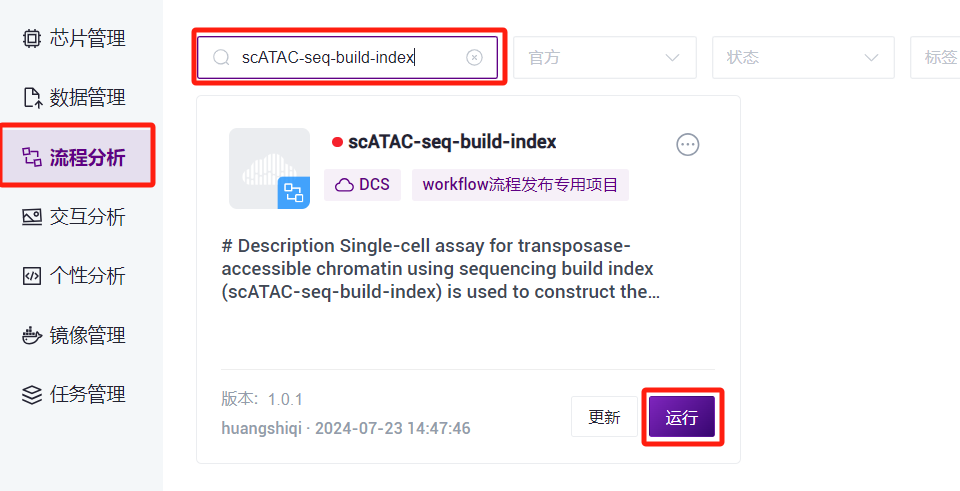
- 点击导航栏**【流程分析】进入流程分析页面,在搜索框输入scATAC-seq-build-index**,点击**【运行】**(图3-3)。
- 选择运行界面输入,输入Entity ID,点击**【下一步】**(图3-4)。
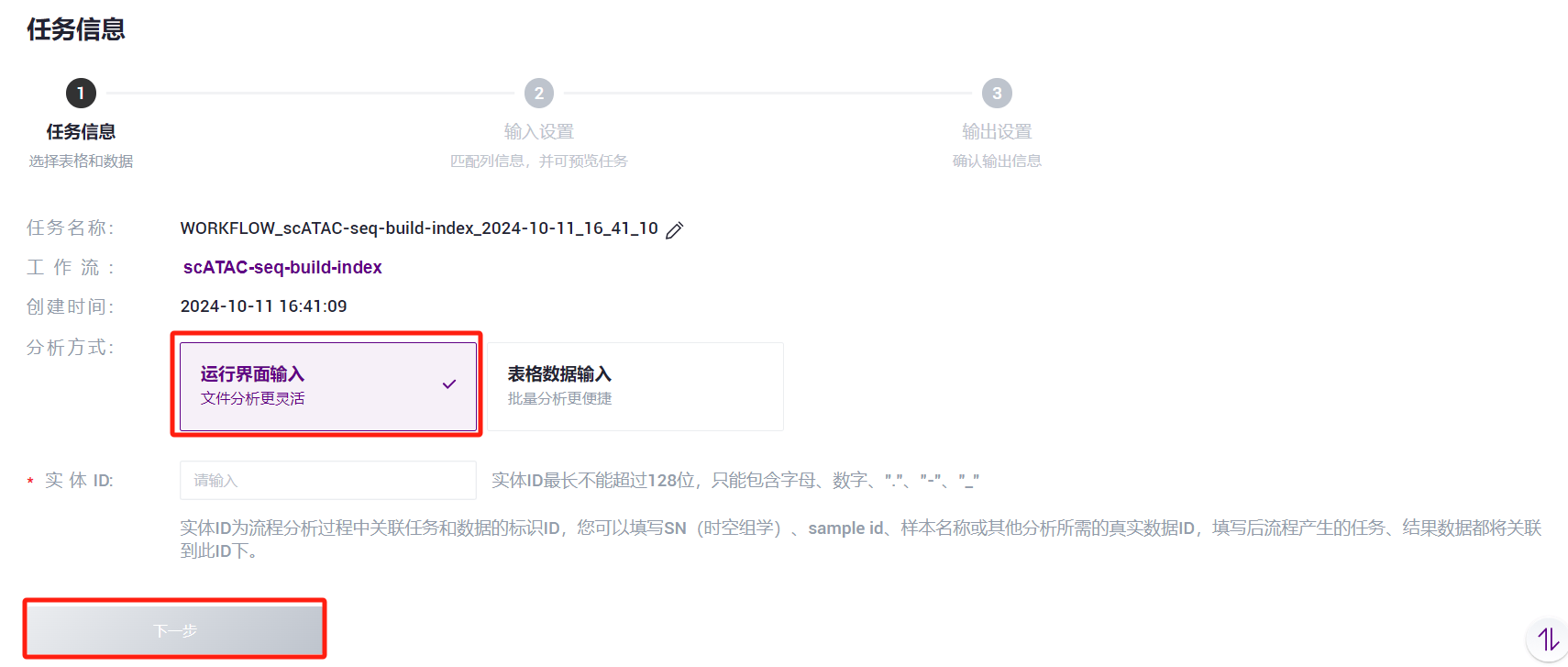
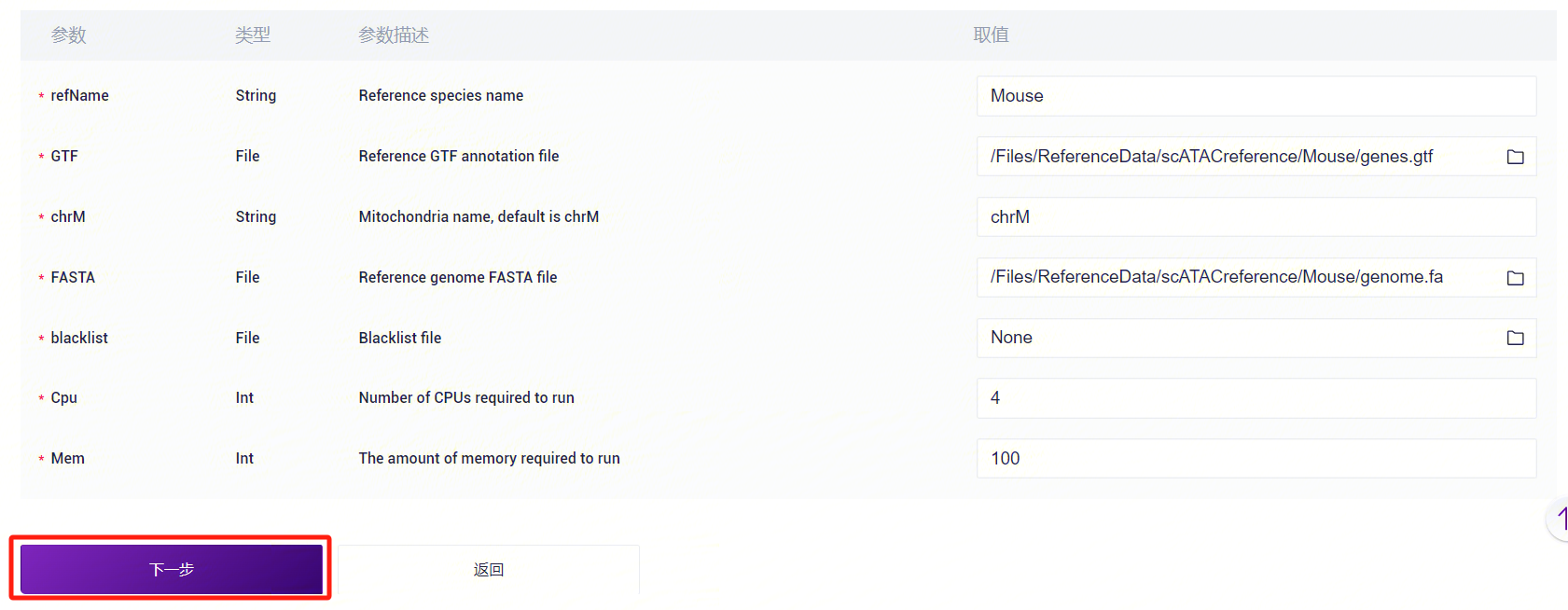
- 录入参考基因组信息,完成后点击**【下一步】**(图3-5)。
注意
参考基因组信息录入参数说明:
- refName:参考基因组的物种名称;
- GTF:基因组注释GTF文件;
- chrM:线粒体名称;
- FASTA:基因组FASTA文件;
- blacklist:Blacklist文件,如没有则填None;
- Outdir:输出文件路径;
- Cpu:运行所需CPU;
- Mem:运行所需内存大小。
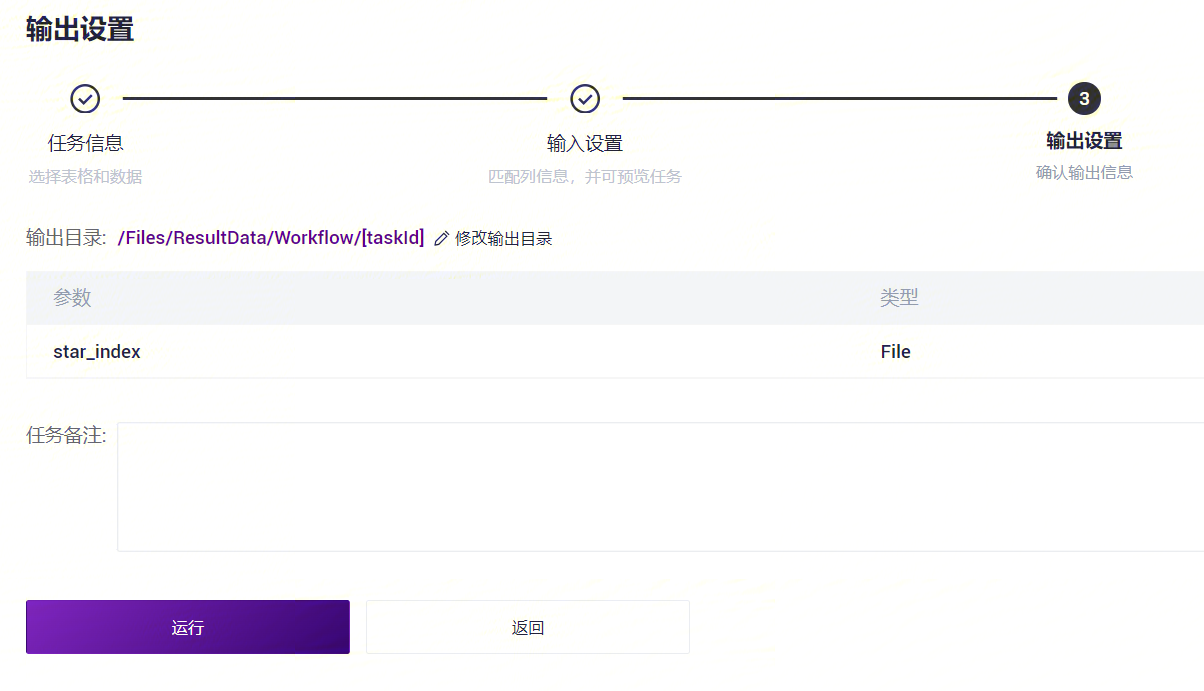
- 点击**【运行】**启动分析(图3-6)。
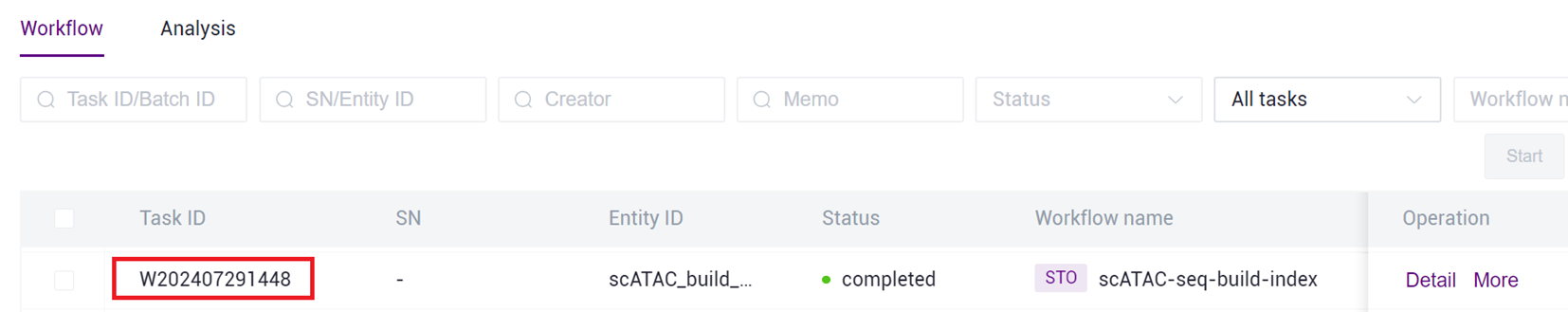
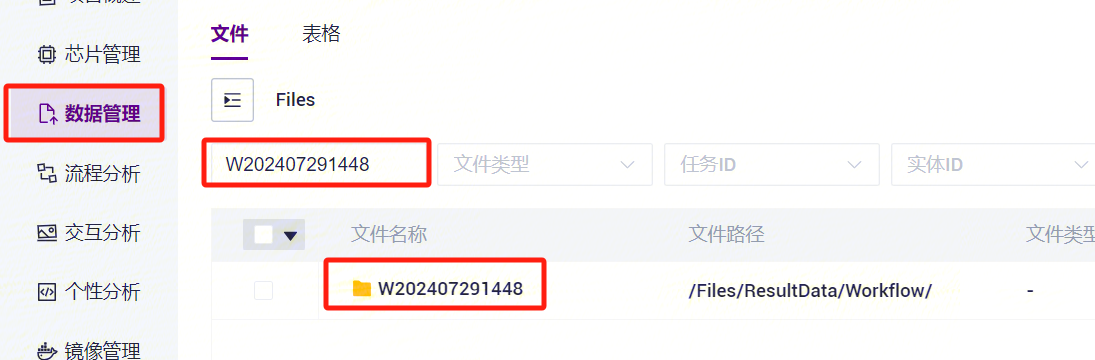
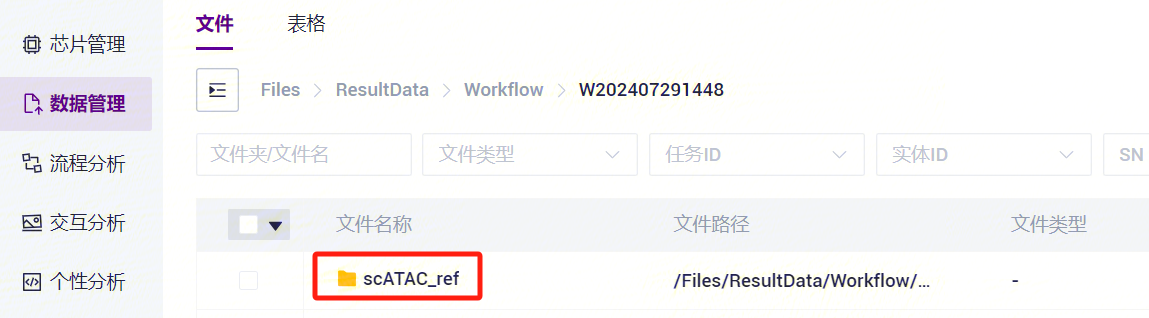
- 任务完成后,Status显示为completed,复制Task ID(图3-7),点击导航栏**【数据管理】,输入Task ID** 进行搜索(图3-8),其中 scATAC_ref 文件将作为参考基因组文件参与scATAC-seq_v3分析(图3-9)。
3.2.3 步骤三:样本信息录入
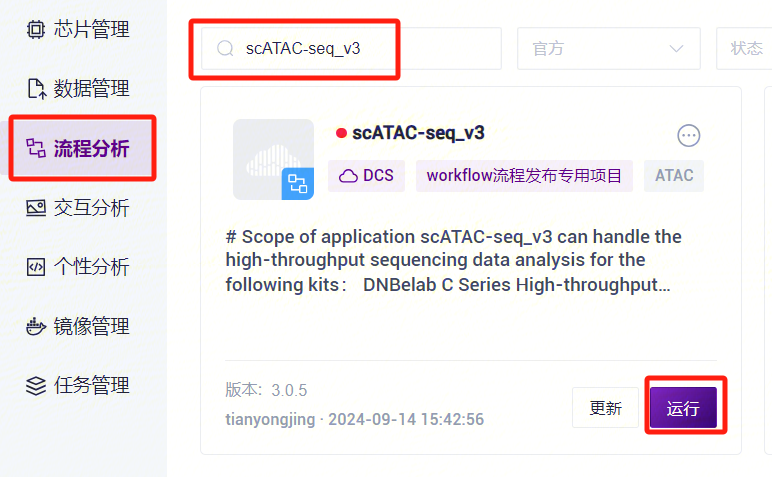
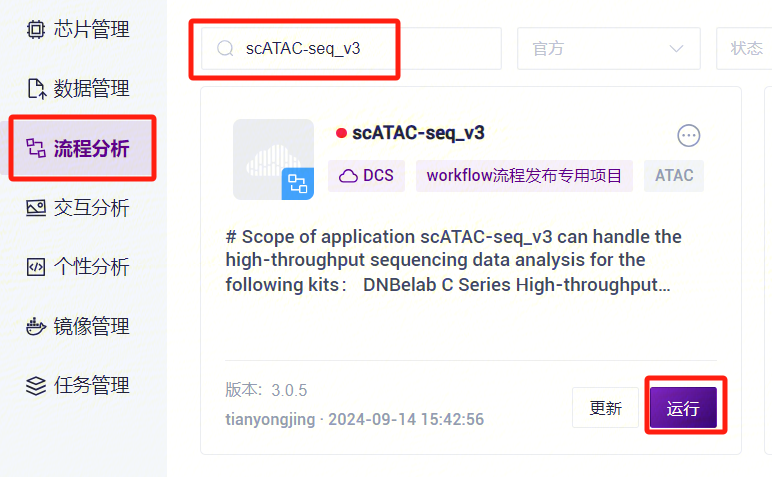
- 点击导航栏**【流程分析】**进入流程分析页面,在搜索框输入 scATAC-seq_v3,点击【运行】(图3-10)。
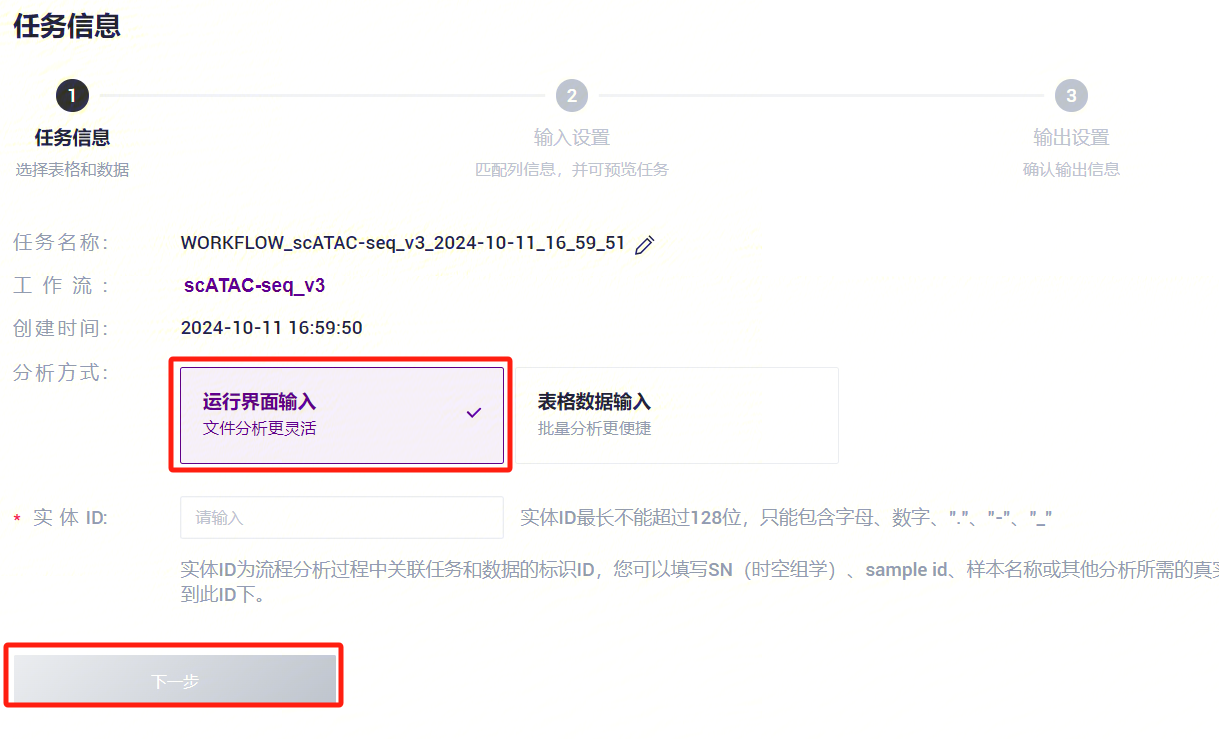
- 选择运行界面输入,输入Entity ID,点击**【下一步】**(图3-11)。
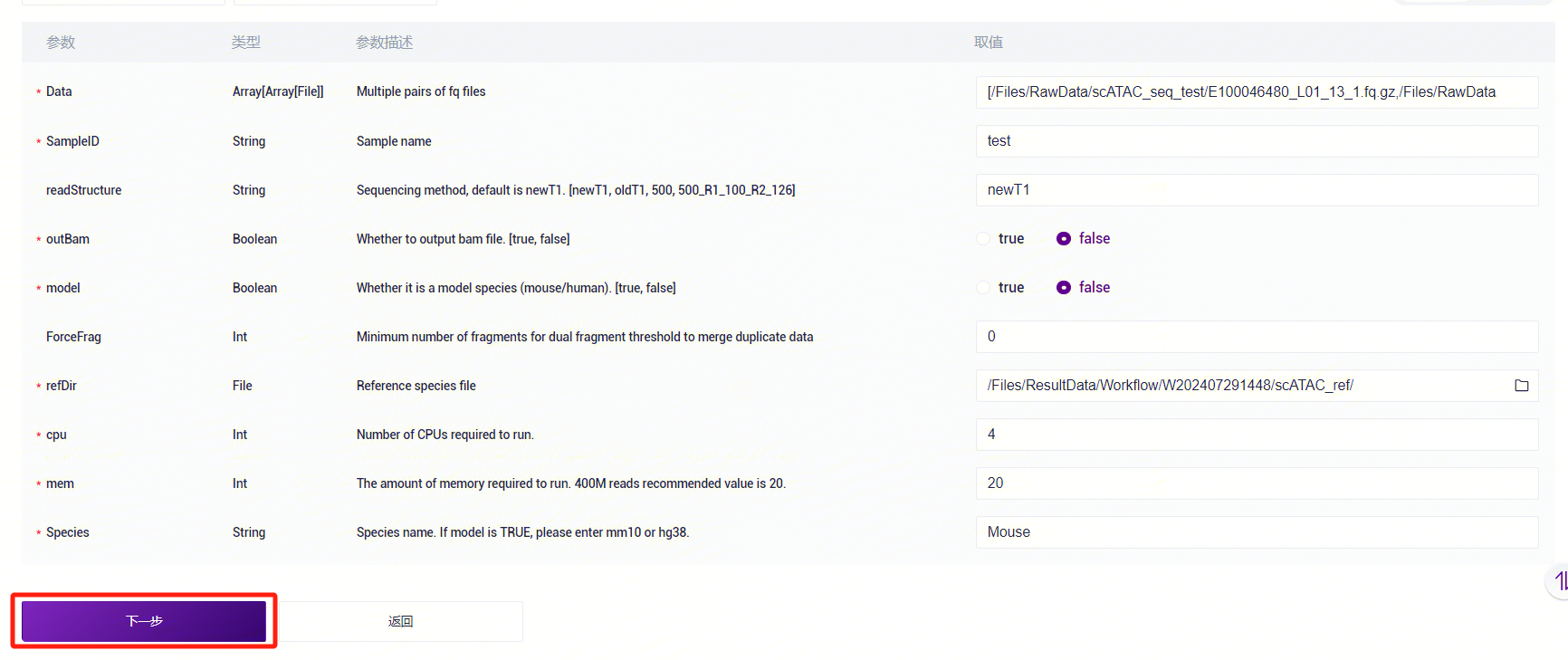
- 录入样本信息,完成后点击**【下一步】**(图3-12)。
注意
样本信息录入参数说明:
Data:FASTQ格式的R1、R2端序列,注意每对FASTQ要先选R1,再选R2;
Outdir:输出文件路径;
SampleID:样本名称,默认与Entity ID一致;
readStructure(选填):测序方法,默认为newT1;
OutBam:是否需要输出bam文件,默认为false;
model:是否为模式生物(mouse/human),默认为false;
ForceFrag(选填):用于合并重复数据的双重片段阈值的最小片段数;
refDir:参考基因组文件,如在步骤二(3.2.2 构建参考基因组)自主构建,选择Task ID文件夹内的scATAC_ref;
BlackList:Blacklist文件,默认为None;
chrMT:线粒体名称,默认为chrM;
genomeSize:基因组大小,与参考基因组构建的结果文件ref.json中genomesize的值一致;
cpp:运行所需CPU大小,默认值为4;
mem:运行所需内存大小,默认值为20;
Species:物种名称,如果model的值为true,则输入mm10或hg38。
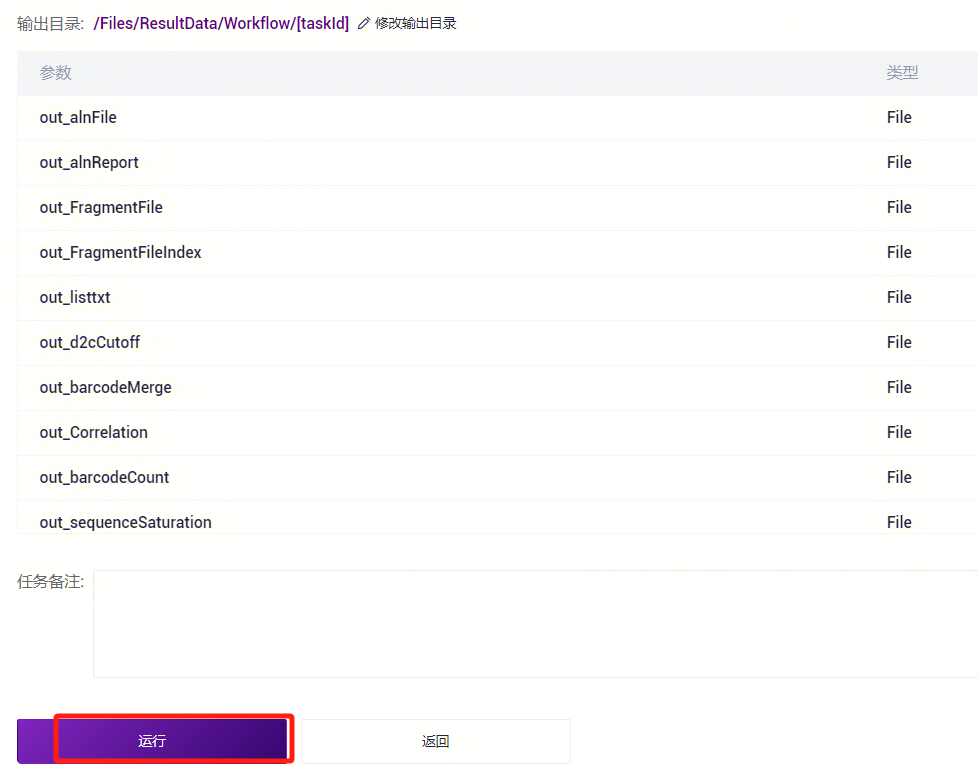
3.2.4 步骤四:启动分析
点击**【运行】**启动分析(图3-13)。
3.3 使用场景二:表格投递
操作共包括五个步骤:上传数据、构建参考基因组(可选)、下载样本模板、填写并导入样本模板、启动分析。完成样本模板导入后,可批量运行任务,当客户在看到任务状态为completed 时,代表任务已完成,即可查看报告部分(详见3.4部分)。
3.3.1 步骤一:上传数据
与使用场景一(手动投递)步骤一一致(详见3.2.1步骤一:上传数据)。
3.3.2 步骤二:构建参考基因组(可选)
与使用场景一(手动投递)步骤二一致(详见3.2.2步骤二:构建参考基因组)。
3.3.3 步骤三:样本信息录入表格下载
- 点击导航栏**【数据管理】,选择【表格】-【下载模板】(如图3-14), 点击【Data model template】**,选择 scATAC-seq_v3 模板下载。
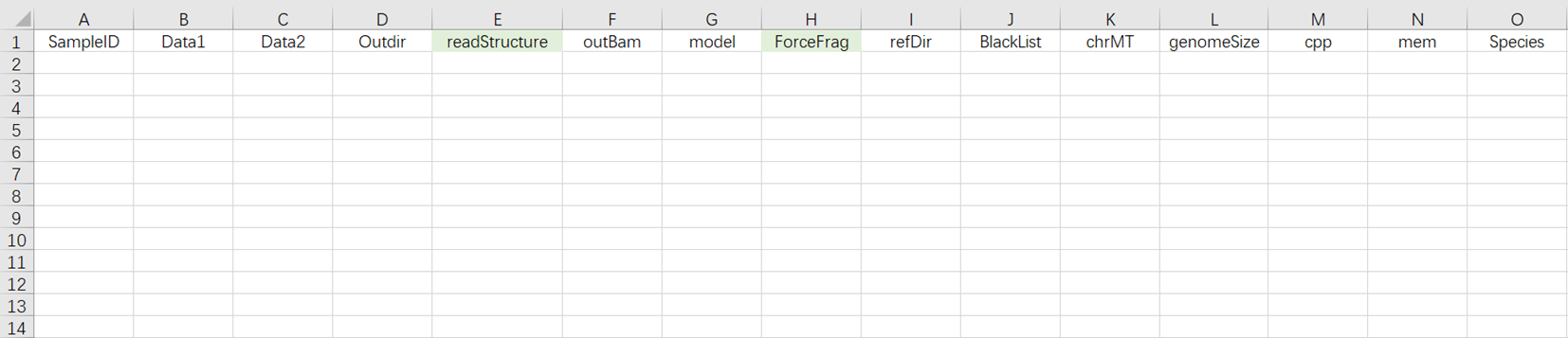
- 打开后scATAC-seq_v3样本模板Excel如图3-15。
3.3.4 步骤四:样本信息导入
- 该使用场景条件下,样本导入表格需填写工作表(图3-15)。该场景表示对已测序完成的样本数据在导入表格后,直接进入分析。
注意
注意事项:
[1] 导入文件路径必须在云平台已存在。
[2] 模板中,除readStructure、ForceFrag外的输入栏为必填项。导入数据中,必填项字段不得为空。
[3] Excel中的SampleID需唯一。
[4] Excel中不能合并单元格,单元格内容前后不能有空格或特殊字符。
[5] 分析样本录入(图3-16):
- Data:FASTQ格式的R1、R2端序列,注意每对FASTQ要先填写R1的绝对路径(Data1处),再填写R2的绝对路径(Data2处);
- Outdir:输出文件路径;
- SampleID:样本名称,默认与Entity ID一致;
- readStructure(选填):测序方法,默认为newT1;
- OutBam:是否需要输出bam文件,默认为false;
- model:是否为模式生物(mouse/human),默认为false;
- ForceFrag(选填):用于合并重复数据的双重片段阈值的最小片段数;
- refDir:参考基因组文件,如在步骤二(3.3.2 构建参考基因组)自主构建,选择Task ID文件夹内的scATAC_ref文件;
- BlackList:Blacklist文件,默认为None;
- chrMT:线粒体名称,默认为chrM;
- genomeSize:基因组大小,与参考基因组构建的结果文件ref.json中的genomesize值一致;
- cpp:运行所需CPU大小,默认值为4;
- mem:运行所需内存大小,默认值为20;
- Species:物种名称,如果model的值为true,则输入mm10或hg38。
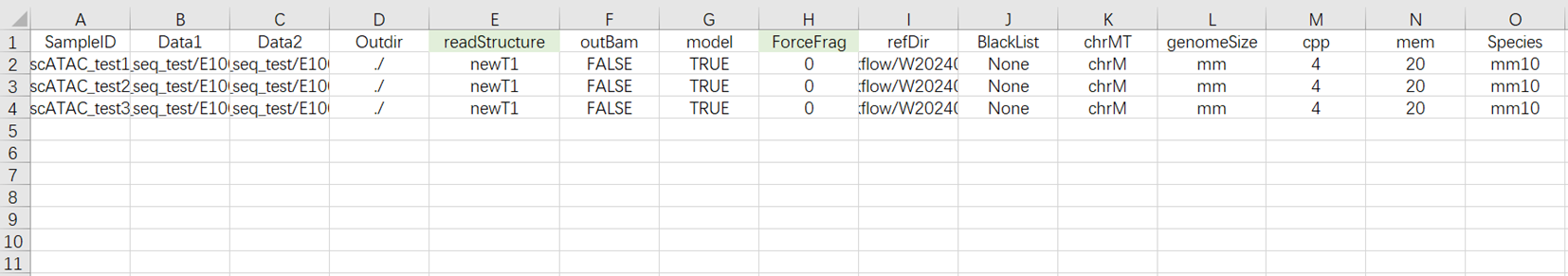
- 配置好样本模板的分析样本录入工作表后,回到**【数据管理】界面,点击【表格】-【+新增表格】**(图3-17)。
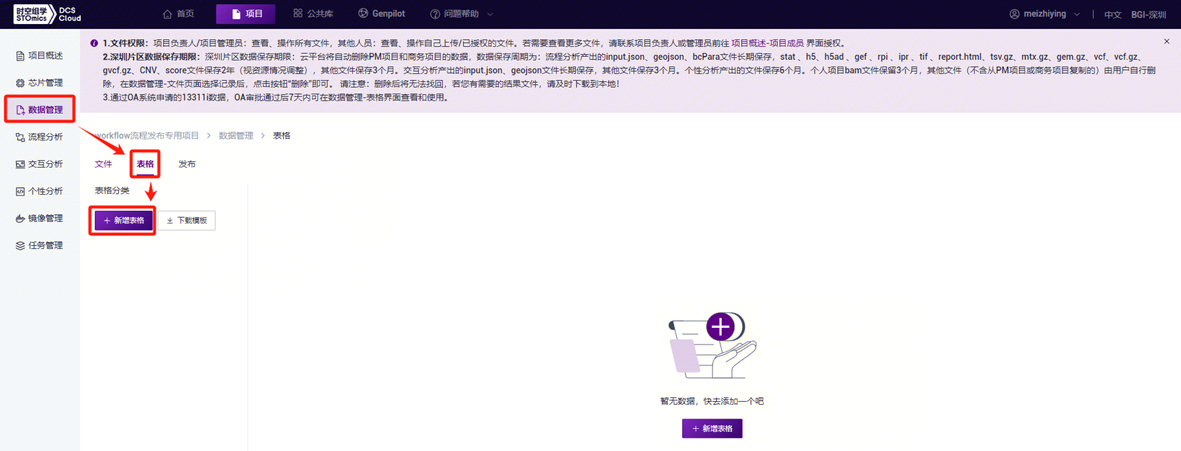
- 点击**【点击上传/拖拽到此区域】浏览并选择已填好样本信息的表格,点击【确定】**(图3-18),上传完成后文件会在目标文件夹中显示。
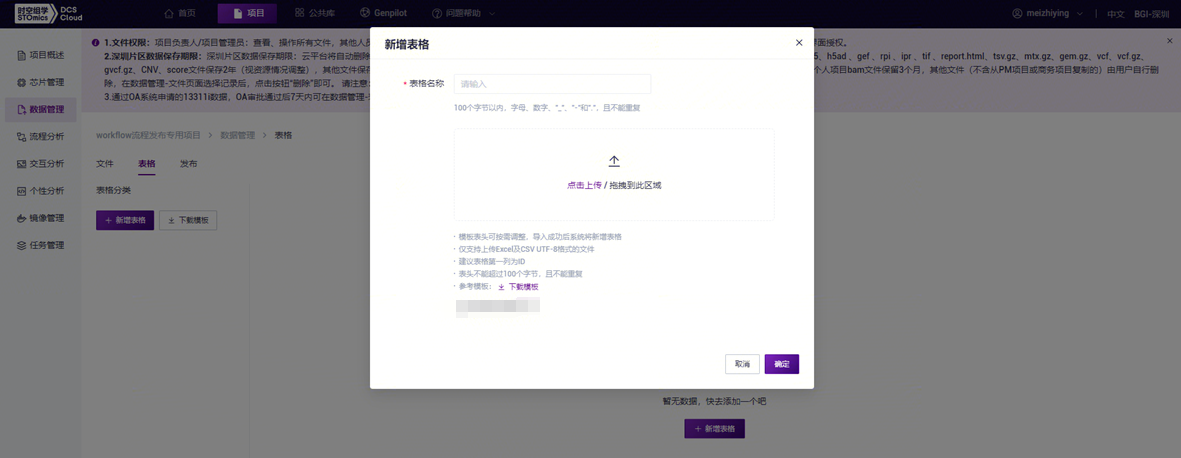
- 点击导航栏**【流程分析】进入流程分析页面,在搜索框输入scATAC-seq_v3****,点击【运行】**(图3-19)。
- 选择表格数据输入,点击 Please select table处,选择在本小节3)中导入的表格,选中所需的行,点击**【下一步】**(图3-20)。
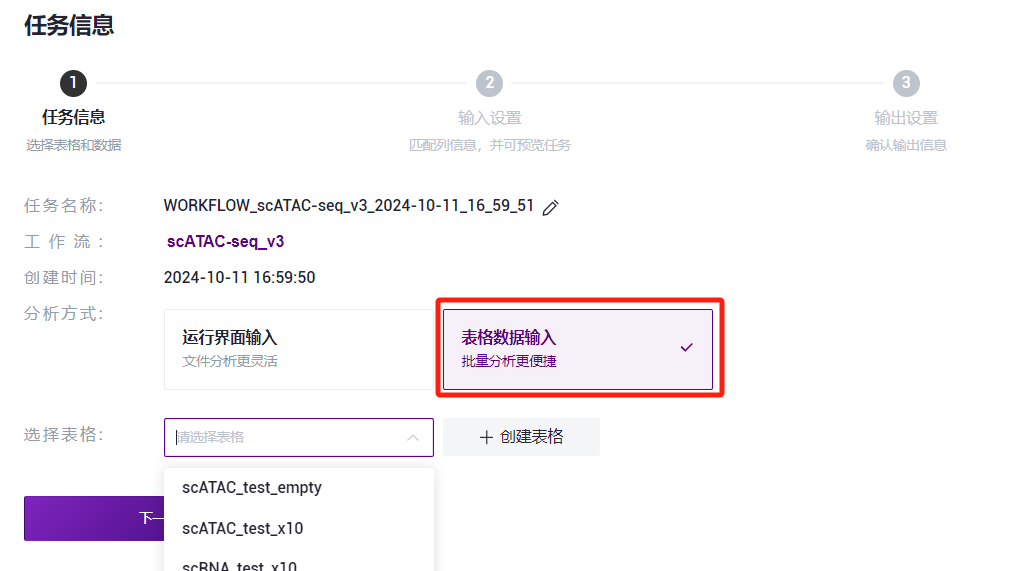
- 在Values处点击并选择对应的值,如Data选取${Data1}和${Data2},注意按顺序选取(如图3-21)。
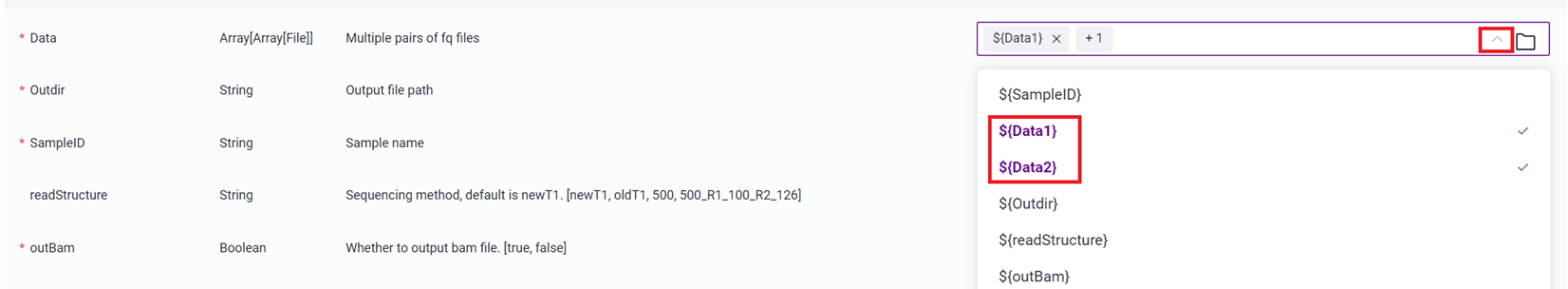
- 录入样本信息,完成后点击**【下一步】**,确保参数设置无误(如图3-22)。
3.3.5 步骤五:启动分析
点击**【运行】**启动分析(图3-23)。
3.4 报告查看及结果文件下载
3.4.1 报告查看
点击导航栏**【任务管理】,当任务状态显示为completed时,代表任务已完成,点击【报告】**即可查看报告部分(如图3-24)。
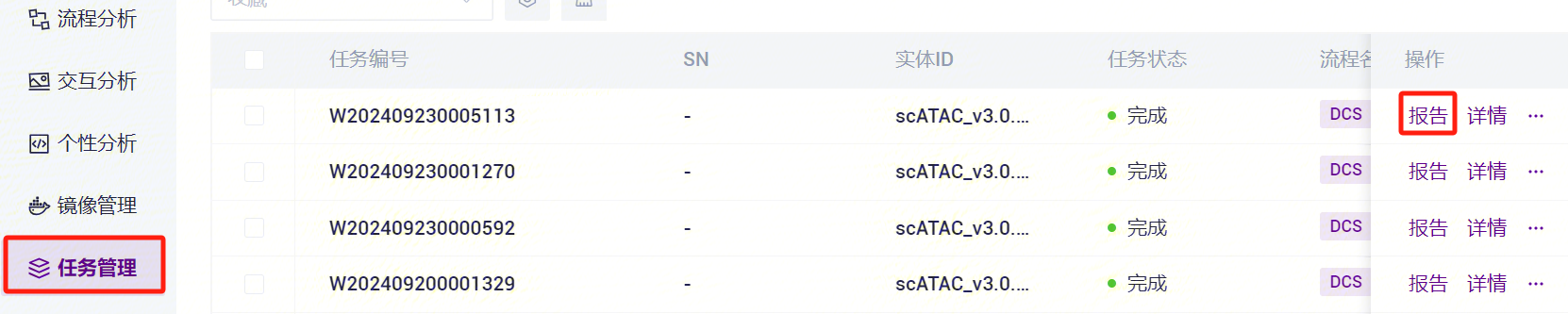
3.4.2 报告下载
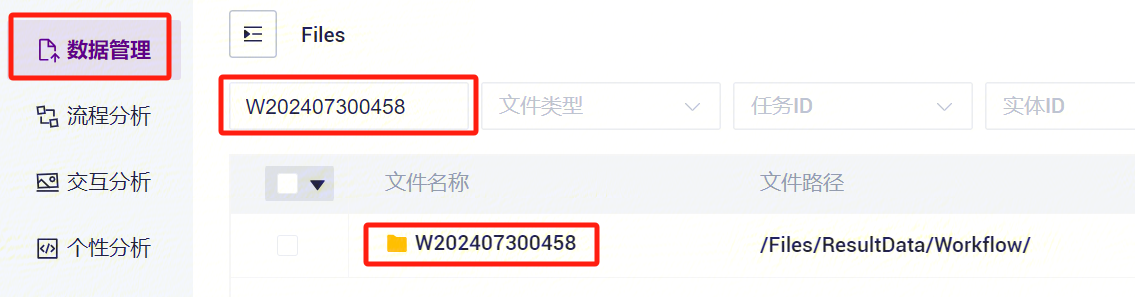
- 点击导航栏**【数据管理】,进入数据管理页面,根据任务的Task ID进行搜索,点击进入Task ID**文件夹(如图3-25)。
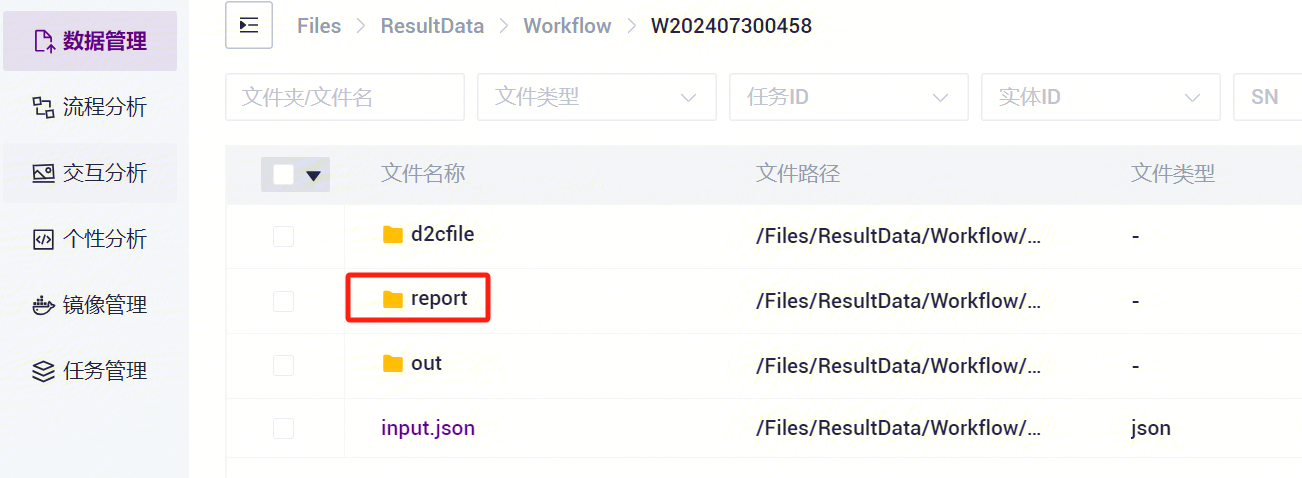
- 点击进入 report 文件夹(如图3-26)。
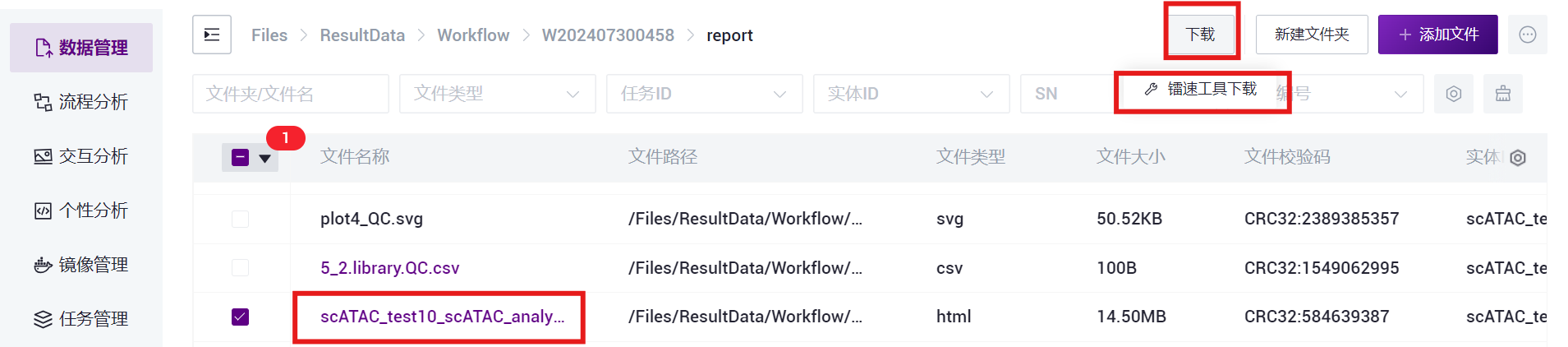
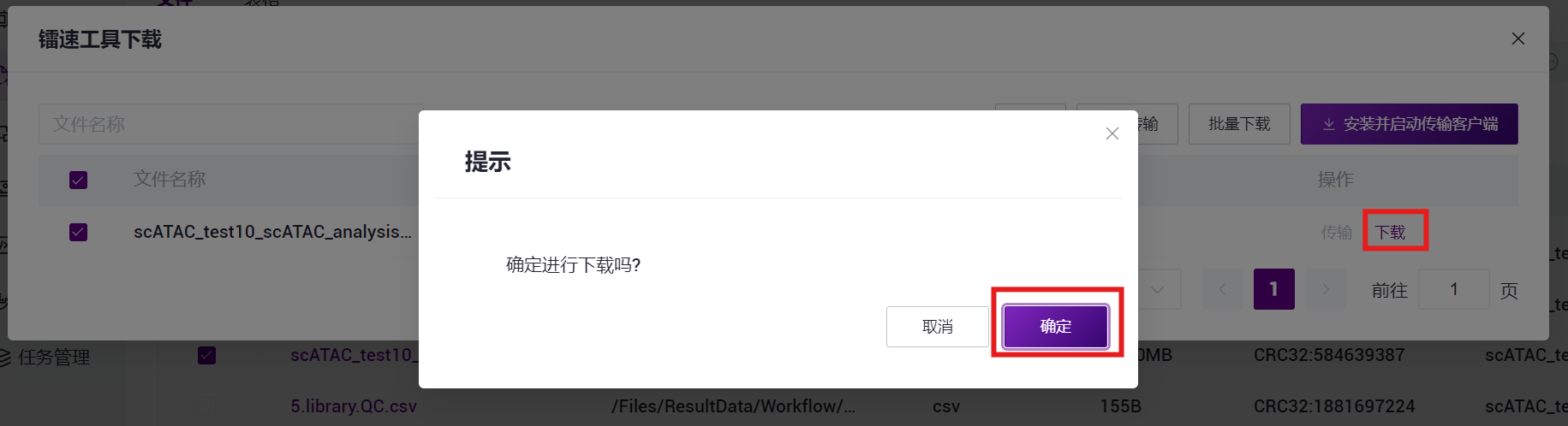
- 选中 report.html 文件,点击**【下载】-【镭速工具下载】**(如图3-27)。
- 点击**【传输】-【下载】-【确定】**,选择目标目录并下载报告(如图3-28)。
4. 第四章 FAQ
- 官方的单细胞流程有哪些,分别对应试剂盒的版本是什么?
scATAC-seq官方维护流程为scATAC-seq_v3,对应试剂盒DNBelab C系列高通量单细胞ATAC文库制备试剂盒套装。
- 构建Reference后gtf文件格式要求是什么?
gtf文件中染色体名称与基因组文件中的染色体名称要一致。