MSAP 使用说明
MSAP使用说明书
版本信息
| 说明书版本 | 流程版本 | 修订日期 | 修订内容摘要 |
|---|---|---|---|
| A1 | V1.0.0 | 2025年7月 | * 首次发布 |
提示:请下载最新版说明书,对照相应版本的流程使用。
产品信息
产品描述
宏基因组是指特定环境中所有微生物群体的基因组集合,包括细菌、真菌、病毒等多种微生物。宏基因组测序能够在无需分离培养的情况下,直接对环境样本中的全部微生物DNA进行高通量测序和分析,是研究微生物多样性、群落结构、功能基因及其与环境互作关系的重要手段。随着测序技术的不断进步和成本的降低,宏基因组测序因其高通量、全面性和灵敏度高等优势,已成为微生物生态学、环境科学、医学等领域的重要研究工具。
宏基因组分析流程(以下简称MSAP)是对环境样本中全部微生物基因组进行测序,通过高质量数据的预处理、去除宿主污染、拼接组装、物种注释、功能注释等步骤,全面解析微生物群落的组成、结构及其功能潜力。宏基因组研究为揭示微生物在生态系统中的作用、环境变化对微生物群落的影响以及微生物与宿主的互作机制提供了强有力的技术支持,目前已广泛应用于土壤、海洋、肠道等多种环境的基础研究、疾病诊断、农业改良和环境治理等领域。
MSAP的核心在于对微生物群落结构和功能的全面解析,利用生物信息学和统计学方法,比较不同环境或处理条件下微生物群落的多样性和功能差异,筛选出与特定环境或表型相关的关键微生物和功能基因,并进一步探究其生态学意义。分析流程包括数据质控、去除宿主序列、拼接组装、物种和功能注释、群落多样性分析及可视化报告生成等环节。通过用户友好的图形化界面实现样本管理和结果展示,支持任务并行化处理,大幅提升分析效率,实现高效、快速的宏基因组数据解读和交付。。
注意事项
本产品仅用于科研用途,不用于临床诊断,使用前请仔细阅读本说明书。
本说明书及其包含的信息为深圳华大生命科学研究院(以下简称深圳华大研究院)的专有保密信息,未经深圳华大研究院的书面许可,任何个人或组织不得全部或部分地对本说明书进行重印、复制、修改、传播或公布给他人。本说明书的读者为终端用户,由深圳华大研究院授权终端用户予以使用。严禁未授权的个人使用本说明书。
深圳华大研究院对本说明书不做任何种类的保证,包括(但不限于)用于特定目的的商业性和合理性的隐含保证。深圳华大研究院已经采取措施,确保本说明书的准确性。但是,深圳华大研究院对错误或遗漏不承担责任,并保留任何对本说明书和流程进行改进以提高其可靠性、功能或设计的权利。
本说明书中的所有图片均为示意图,图片内容可能与使用界面有细微差异,请以实际使用界面为准。
产品介绍
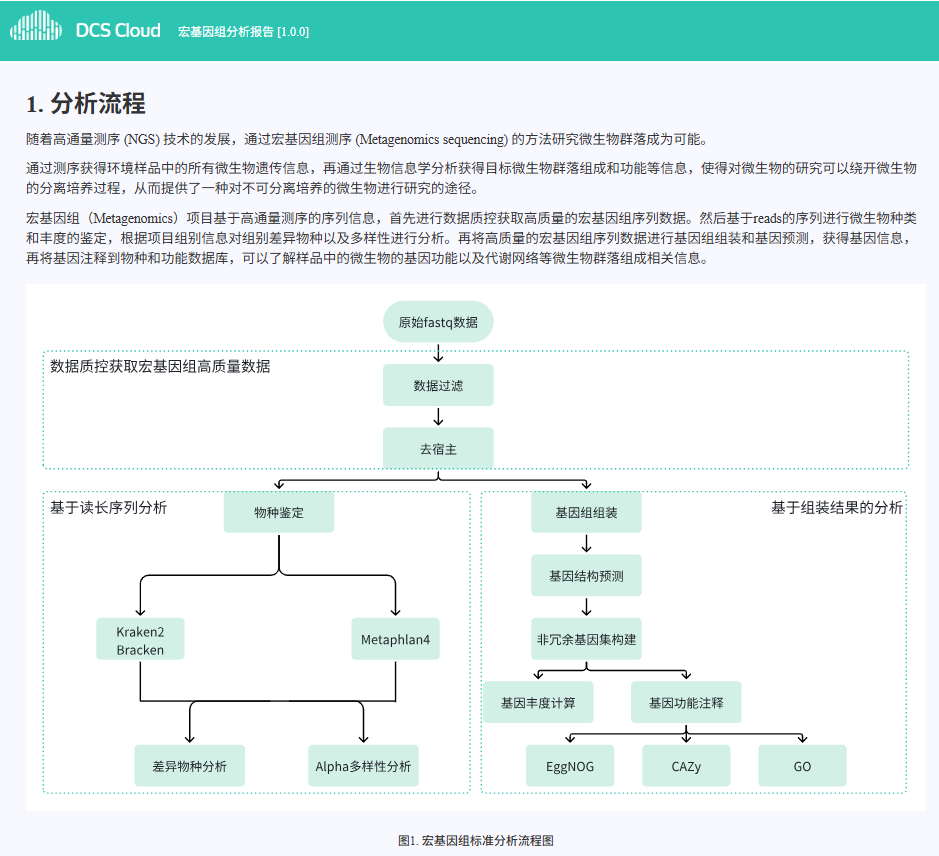
分析流程
MSAP分析流程是基于DCS云平台系统开发的一款自动分析流程,其中包括数据过滤、去除宿主序列、物种鉴定、差异物种分析、Alpha多样性分析、基因组组装、基因结构预测、非冗余基因集构建、基因丰度计算、基因功能注释以及报告生成:
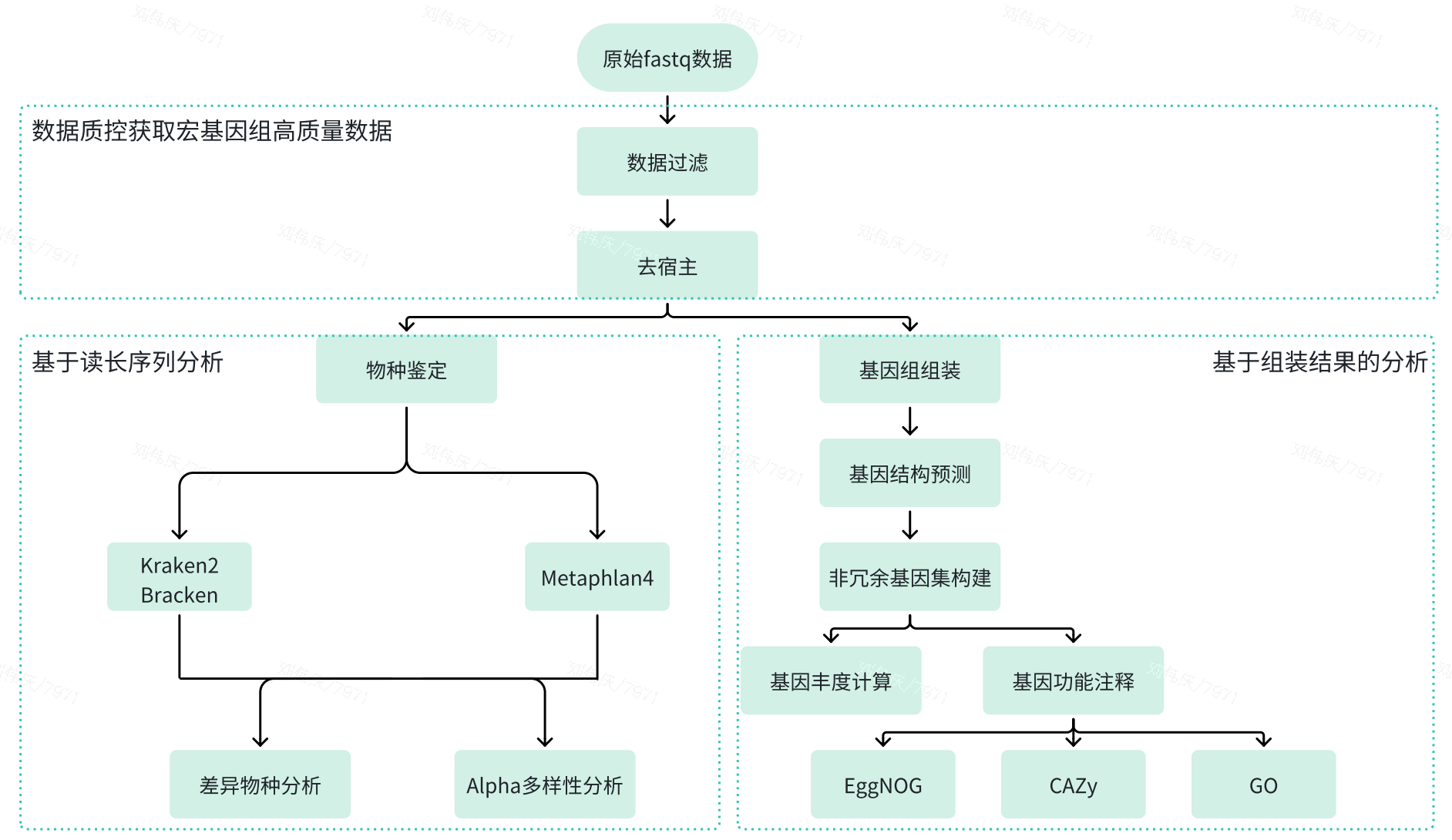
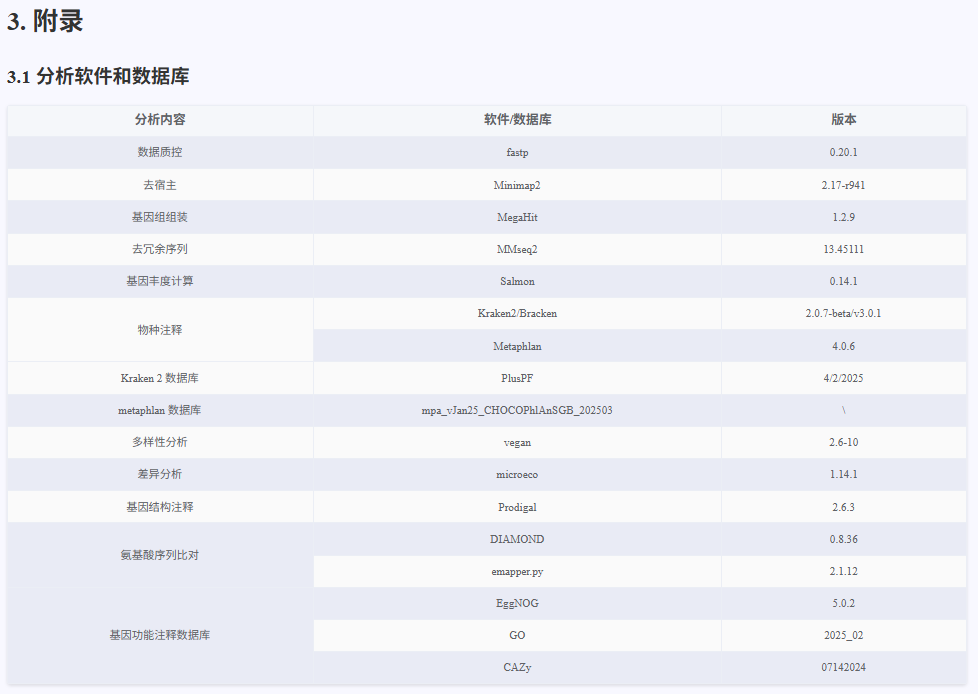
数据过滤
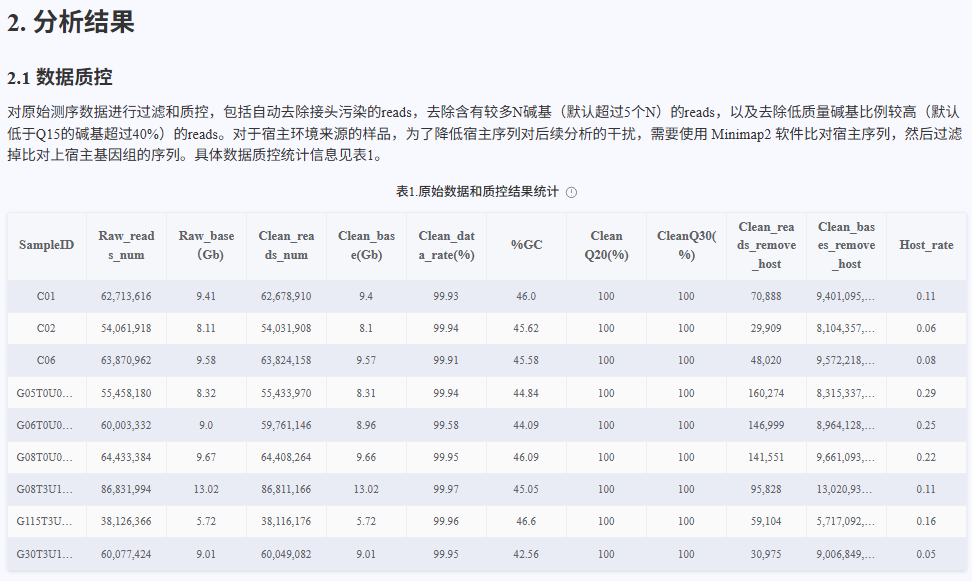
使用 fastp 对原始测序数据进行质量控制和过滤,包括低质量序列过滤、接头序列去除、长度筛选等操作。通过 fastp 处理后,输出质控前后的测序质量统计信息,包括碱基质量分布、序列长度分布、GC含量分布等。过滤后获得的 clean data(高质量数据)将用于后续的比对和分析,确保分析结果的准确性和可靠性。
去除宿主序列
使用 Minimap2 软件将过滤后的 clean reads 与宿主基因组序列进行比对,通过识别并去除与宿主基因组匹配的序列,从而获得去除宿主污染的微生物基因组数据。比对完成后,输出比对结果文件,并统计去除宿主序列前后的数据量,为后续的宏基因组分析提供高质量的非宿主 clean data。
物种鉴定
使用 Kraken2 和 Bracken(或 MetaPhlAn4)对去除宿主序列后的 clean data 进行宏基因组物种鉴定。Kraken2 是一款基于 k-mer 的高效宏基因组分类工具,能够快速准确地对微生物序列进行分类注释。Bracken 可在 Kraken2 分类结果的基础上进一步优化物种丰度的定量分析。MetaPhlAn4 则基于特异性标志基因进行物种注释和丰度估算,适用于高分辨率的微生物群落结构分析。
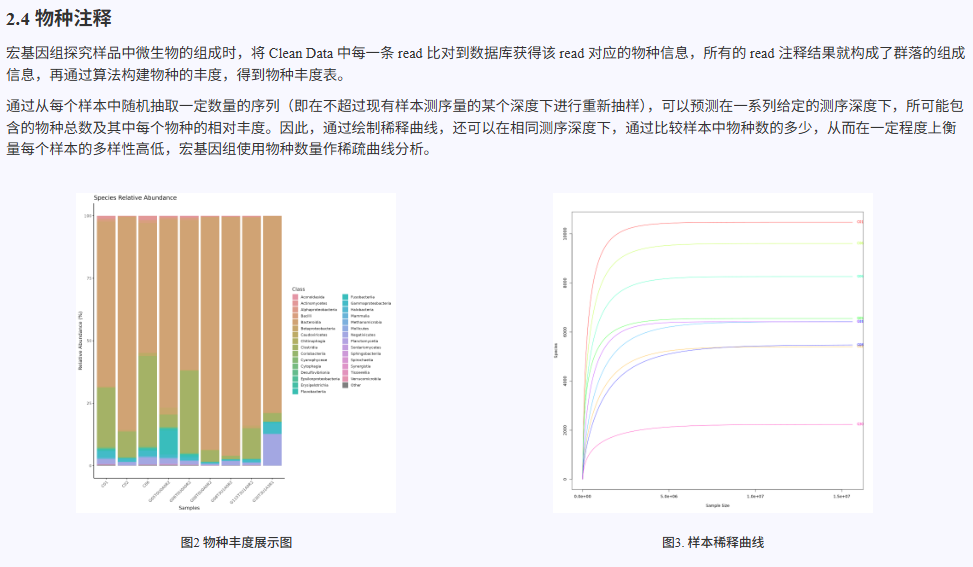
通过上述工具对样本进行物种鉴定后,获得各样本的物种丰度矩阵,全面反映微生物群落的组成和结构。分析流程还包括对物种丰度数据的可视化展示,直观展示不同样本间的微生物多样性和丰度分布。同时,基于物种丰度矩阵生成稀释曲线,评估测序深度对物种多样性捕获的影响,判断测序数据是否满足后续分析需求。
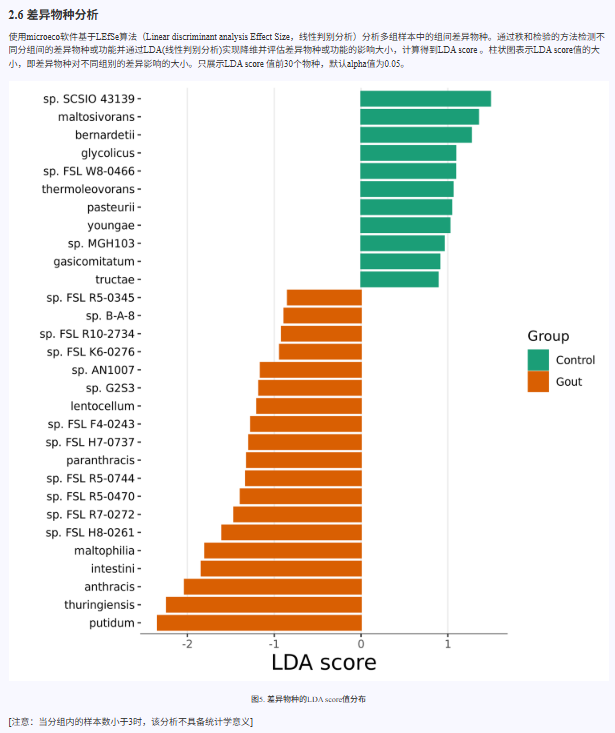
差异物种分析
使用 microeco 软件基于 LEfSe 算法(Linear discriminant analysis Effect Size,线性判别分析)对多组样本进行组间差异物种分析。LEfSe 算法能够结合统计检验和 LDA(线性判别分析),筛选出在不同分组间具有显著差异的物种,并评估其对分组区分的贡献。通过 microeco 对物种丰度数据进行 LEfSe 分析,识别出各组间具有统计学意义的差异物种,并计算每个物种的 LDA score。分析结果以柱状图形式展示 LDA score 值排名前 30 的物种,直观反映不同分组间的优势物种及其区分能力。
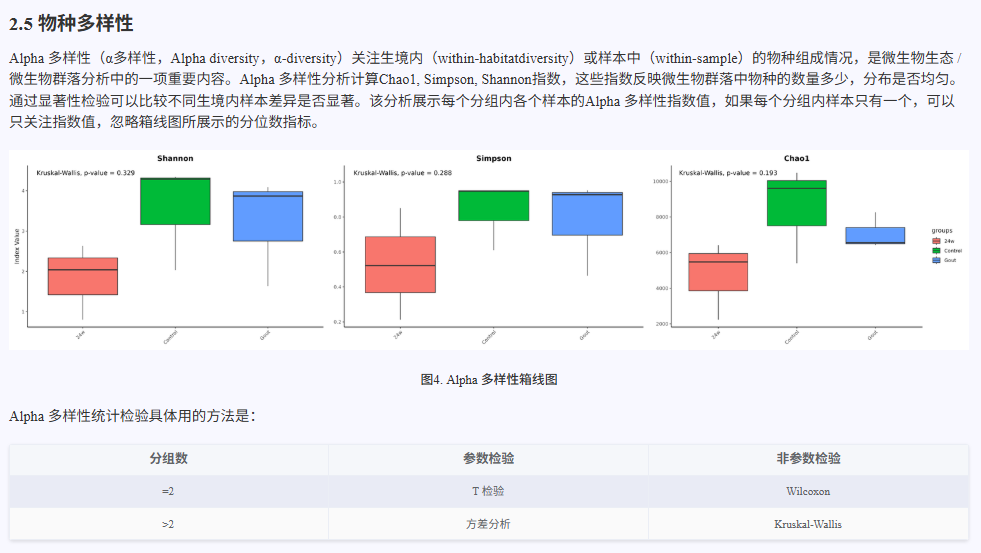
Alpha多样性分析
使用 vegan 软件包对样本进行 Alpha 多样性分析,计算每个样本的 Chao1 指数(反映物种丰富度)、Simpson 指数和 Shannon 指数(反映物种多样性和均匀度)。分析结果能够全面展示各样本的物种多样性水平,为后续群落结构比较和生态学分析提供基础数据。
基因组组装
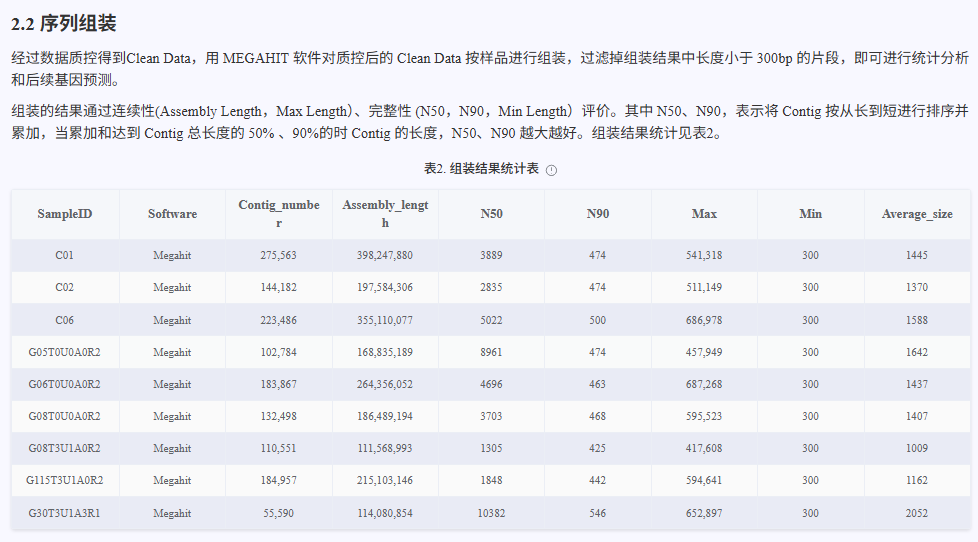
使用 MEGAHIT 软件包对去除宿主序列后的 clean data 按样品进行组装。组装完成后,对结果进行长度筛选,过滤掉长度小于 300bp 的片段,仅保留长度大于等于 300bp 的 contig 进行后续统计分析和基因预测。
基因结构预测
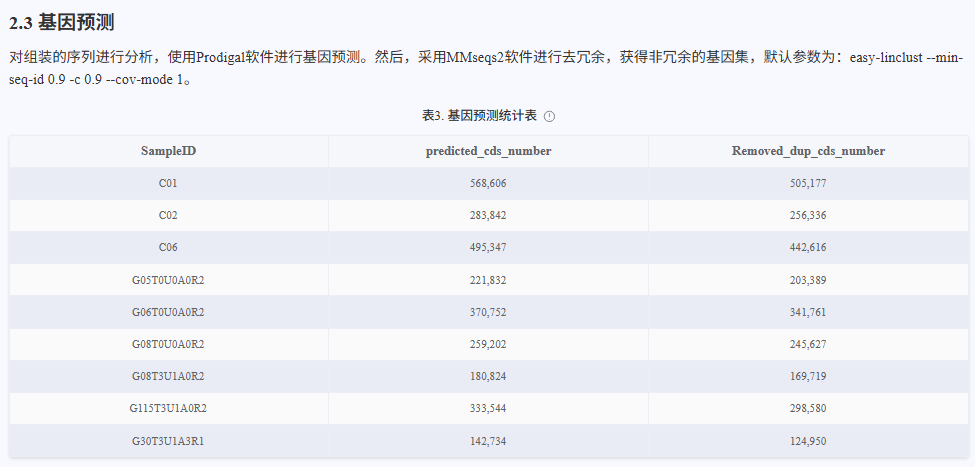
使用 Prodigal 软件包对组装后的基因组序列进行基因结构预测,识别基因的起始和终止位置,预测编码区(CDS)及其对应的蛋白序列。通过对组装后的 contig 进行基因结构预测,获得每个样本的基因注释信息和蛋白序列,为后续的功能注释和多样性分析提供基础数据。
非冗余基因集构建
使用 MMseqs2 软件包对预测得到的基因结构进行去冗余处理,生成非冗余的基因集。通过对所有样本的基因预测结果进行聚类,合并同源或高度相似的基因,获得代表性非冗余基因集(Unigene set),为后续的功能注释、丰度分析和多样性研究提供高质量的基因资源。
基因丰度计算
使用 Salmon 软件包对非冗余基因集进行丰度定量分析,获得每个样本中非冗余基因的表达丰度。通过 Salmon 对 clean data 与非冗余基因集进行比对,生成各样本的基因丰度矩阵,全面反映微生物群落的功能组成和丰度分布。结果中包括对丰度数据的可视化展示,直观展示不同样本间的基因丰度差异。
基因功能注释
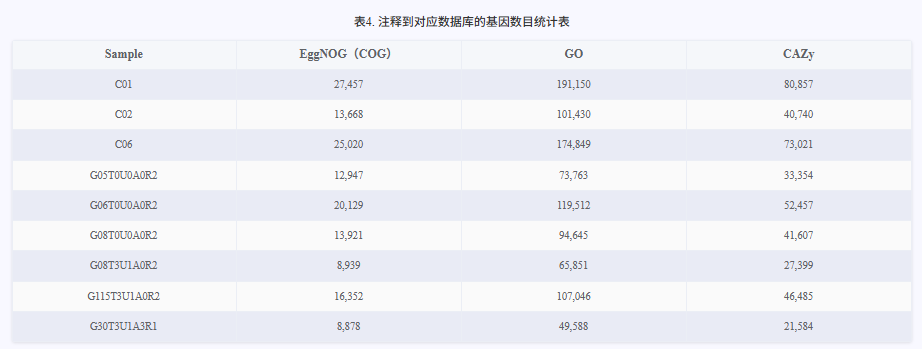
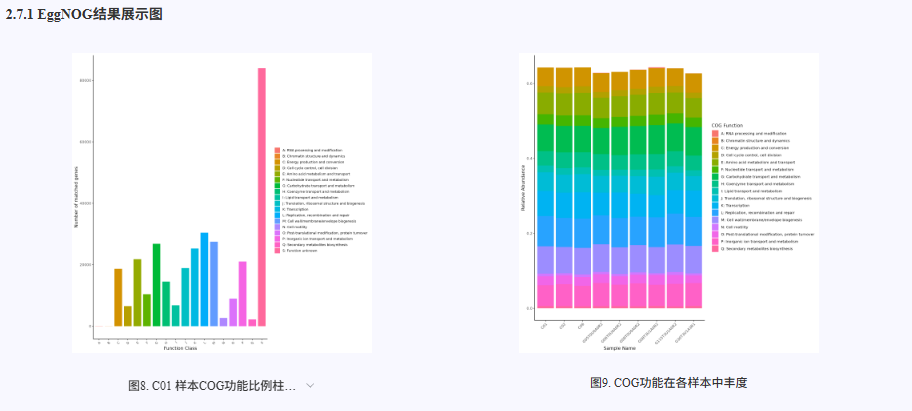
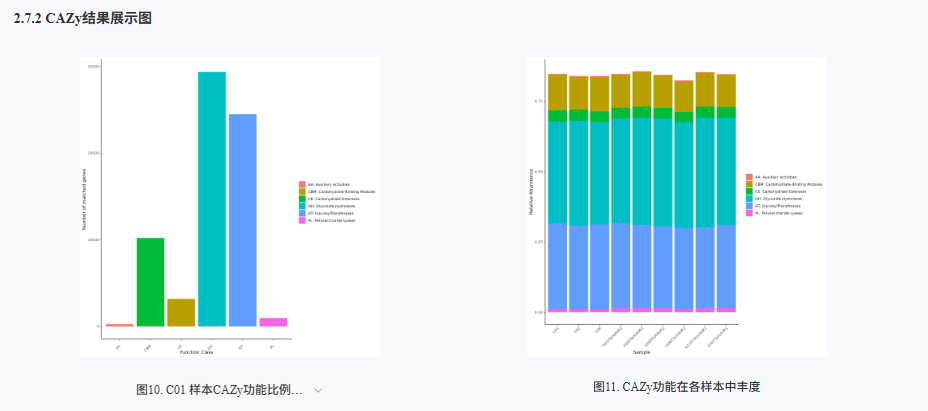
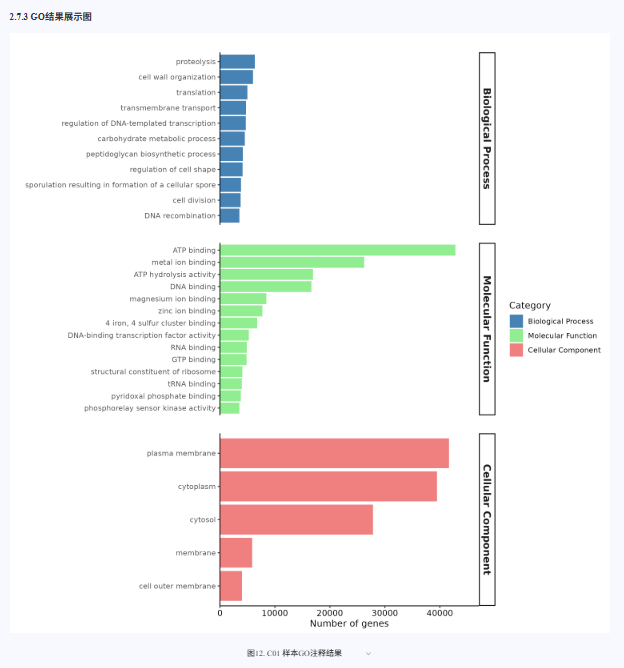
对非冗余蛋白质序列进行基因功能注释,分别采用多种权威数据库和分析工具。使用 Diamond 软件基于 CAZy 和 GO 数据库对蛋白质序列进行快速比对和功能注释,识别与碳水化合物活性酶及基因本体(Gene Ontology)相关的功能信息。使用 emapper.py 工具基于 EggNOG 数据库进行注释,全面解析蛋白质的进化关系和功能类别。
报告生成
综合分析结果,整理汇总成HTML报告。
使用说明书
MSAP分析流程通过DCS云平台系统进行样本输入及报告输出的全流程管理。下面具体介绍基于DCS云平台系统使用MSAP分析流程的操作指南。
指南概述
概述
本章介绍如何使用MSAP分析流程进行分析。在使用之前,请认真阅读并理解其中内容,保证能够正确使用MSAP分析流程。
使用场景一:手动投递
操作共包括四个步骤:上传数据、构建宿主参考基因组(可选)、样本信息录入、启动分析。完成样本信息录入后,运行任务,当客户在看到任务状态为![]() completed 时,代表任务已完成,即可查看报告部分(详见3.4部分)。
completed 时,代表任务已完成,即可查看报告部分(详见3.4部分)。
步骤一:上传数据
- 点击导航栏【Data】,进入数据管理页面,进入目标文件夹,点击右上角【+Add files】-【Toolupload】上传数据(图3.1)

- 点击**【Upload】浏览并选择所需文件(图3.2),上传完成后文件会在目标文件夹中显示(如首次上传,则需点击【Install and start transport client】安装所需工具)

步骤二:构建宿主参考基因组(可选)
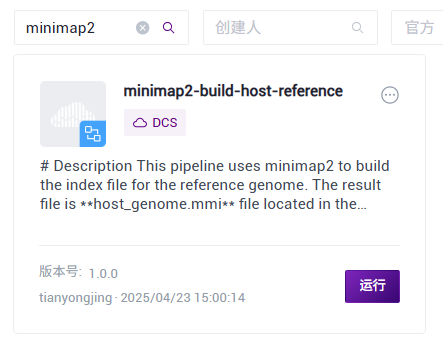
- 点击导航栏【Workflow】进入流程分析页面,在搜索框输入minimap2-build-host-reference,点击【Run】(图3.3)
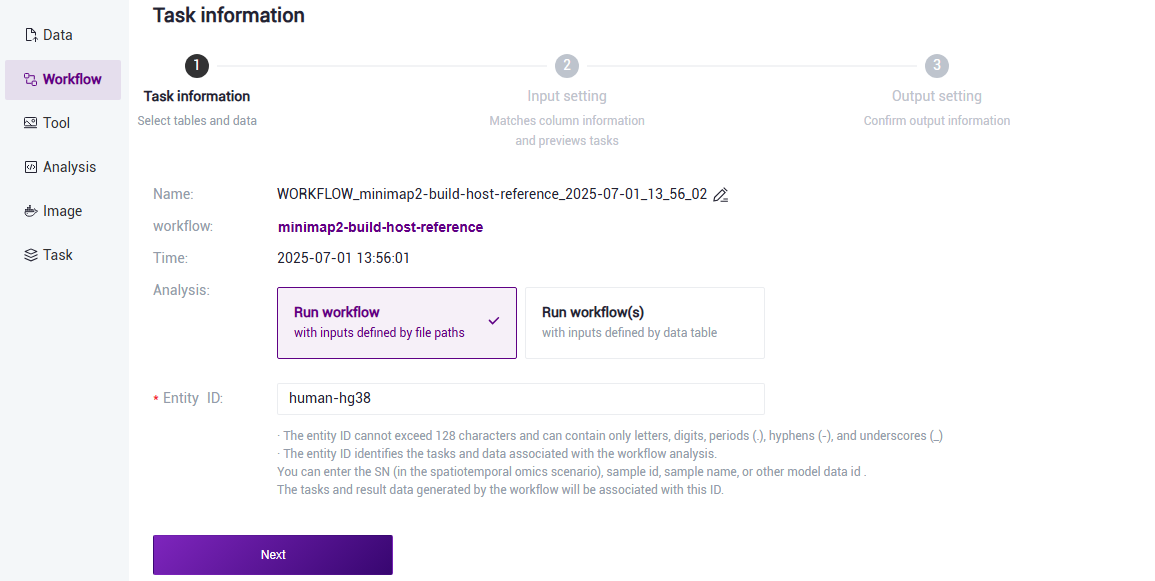
- 选择Run workflow,输入Entity ID,点击【Next】(图3.4)
- 录入参考基因组信息,完成后点击【Next】(图3.5)
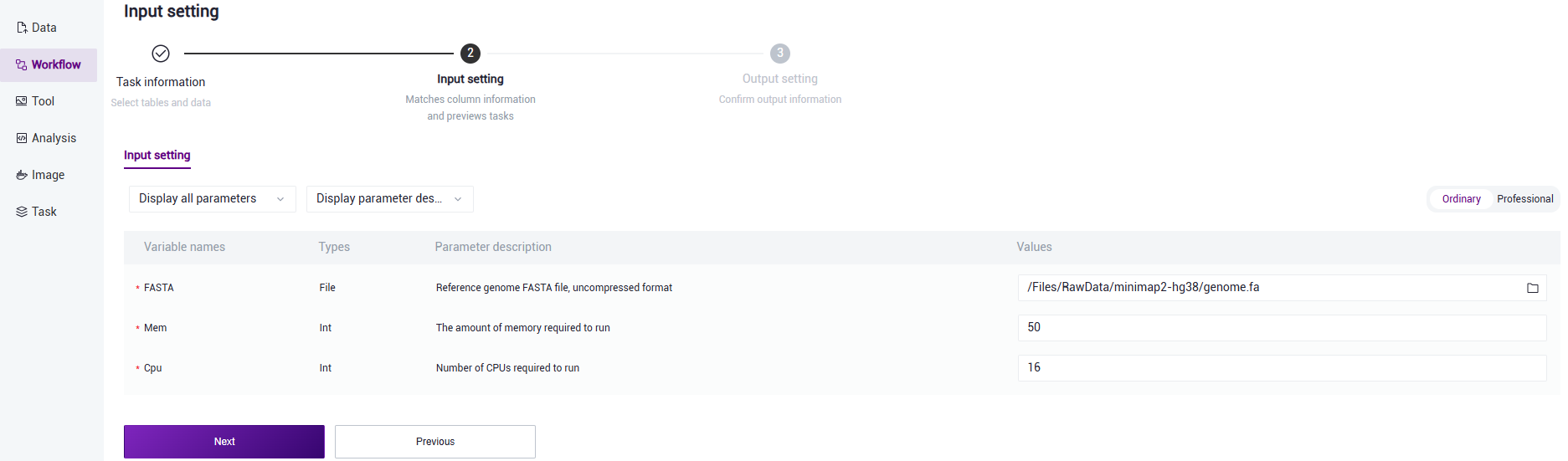
参考基因组信息录入参数说明:FASTA:基因组FASTA文件,不支持压缩格式;Mem:申请任务运行内存;Cpu:申请任务运行线程数。
- 点击【Run】启动分析(图3.6)

- 任务完成后,Status显示为
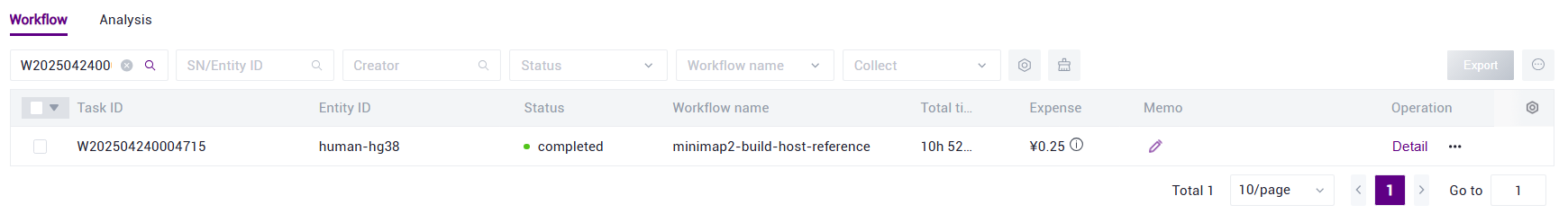
 completed,复制 Task ID(图3.7),点击导航栏【Data】,输入Task ID 进行搜索,其中 star_index 文件作为参考基因组文件夹(图3.8)。
completed,复制 Task ID(图3.7),点击导航栏【Data】,输入Task ID 进行搜索,其中 star_index 文件作为参考基因组文件夹(图3.8)。

步骤三:样本信息录入
- 点击导航栏【Workflow】进入流程分析页面,在搜索框输入MSAP,点击【Run】(图3.10)

- 选择Run workflow,输入Entity ID,点击【Next】(图3.11)
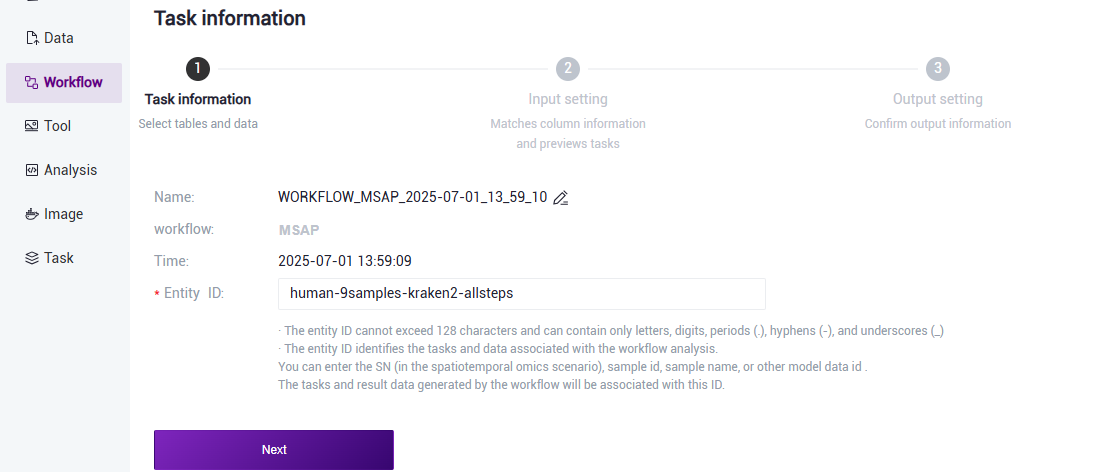
- 录入样本信息,完成后点击**【Next】(图3.12)
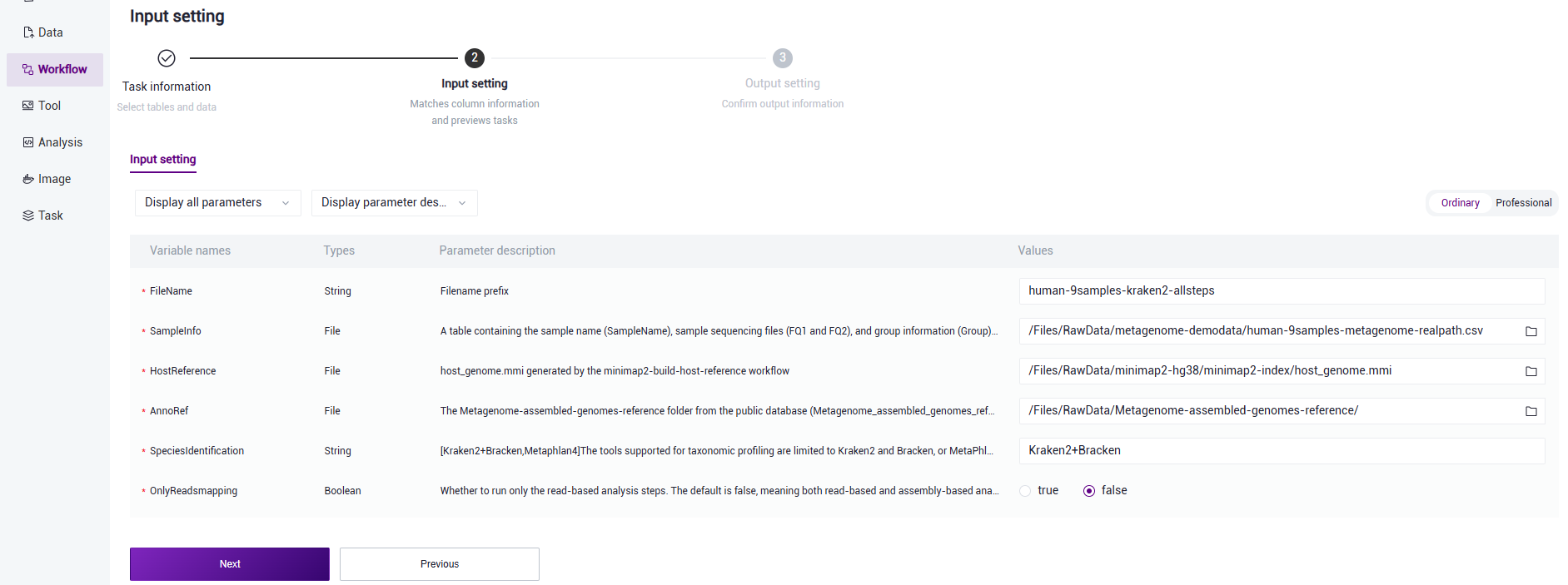
样本信息录入参数说明
FileName:输出文件名前缀;
SampleInfo:包含样本名(SampleName)、样本测序文件(FQ1和FQ2)和分组信息(Group)的表格; 由用户在excel中填写,保存为csv格式,上传至数据管理;其中,样本名由英文字母、数字和下划线组成,相同的样本名视为同一个样本,所有FQ文件将会合并分析;样本测序文件路径要求是绝对路径而非相对路径,双端测序数据分别填写FQ1和FQ2,不支持单端测序数据;DCS云平台绝对路径如下图:
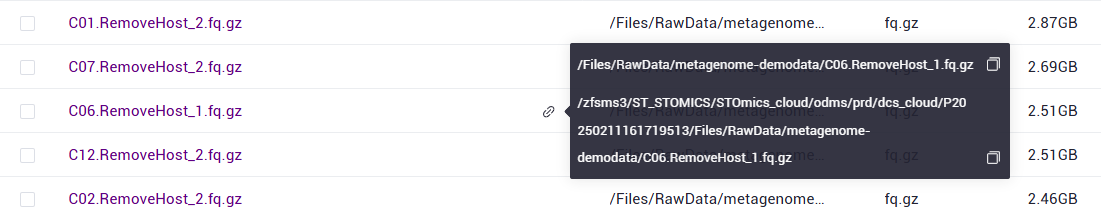 鼠标悬浮于文件旁边的🔗符号,复制红框中文件路径,输入至表格中的FQ1或FQ2分组建议存在生物学重复,由英文字母、数字和下划线组成;
鼠标悬浮于文件旁边的🔗符号,复制红框中文件路径,输入至表格中的FQ1或FQ2分组建议存在生物学重复,由英文字母、数字和下划线组成;

HostReference:宿主的参考基因组文件,如在步骤二(3.2.2 构建宿主参考基因组)自主构建,选择Task ID文件夹内的minimap2-index/host_genome.mmi文件;
AnnoRef:公共数据库中的Metagenome-assembled-genomes-reference文件夹(需要先从公共库Metagenome_assembled_genomes_reference复制到项目的数据管理),用于物种和功能注释;
SpeciesIdentification:用于物种鉴定的工具,仅支持MetaPhlan4或Kraken2,只允许填写Kraken2+Bracken或者MetaPhlan4,默认使用Kraken2+Bracken。
OnlyReadsmapping:是否仅运行基于读长分析步骤,默认为false即运行基于读长分析和基于组装分析。
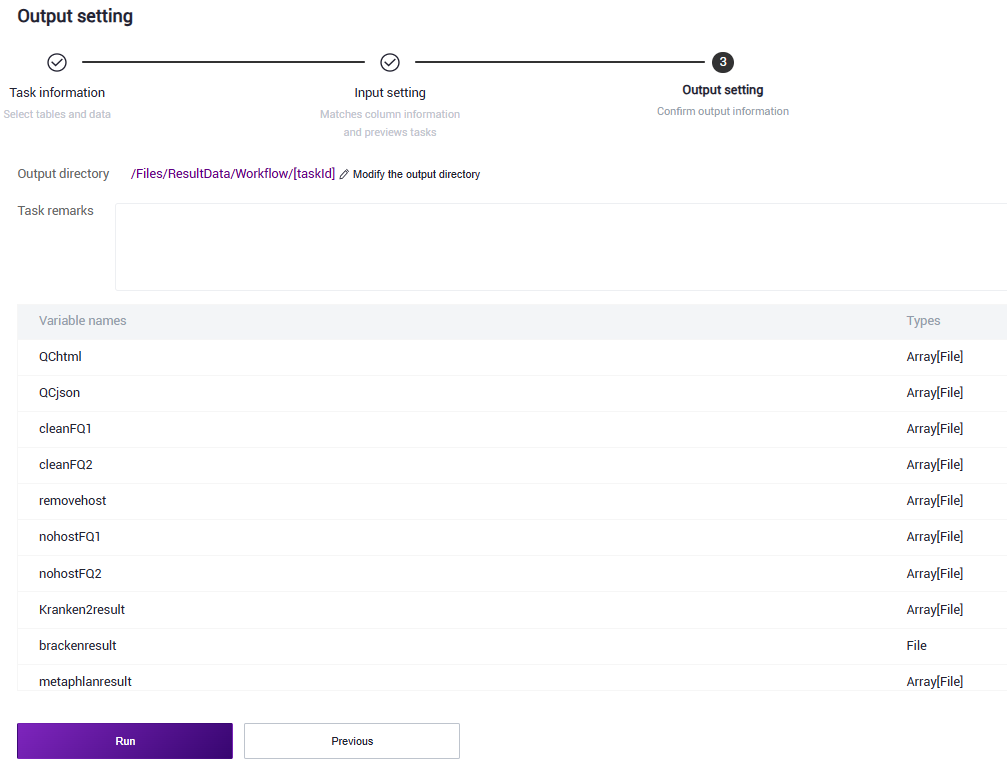
步骤四:启动分析
点击【Run】启动分析(图3.13)
使用场景二:表格投递
操作共包括五个步骤:上传数据、构建宿主参考基因组(可选)、下载样本模板、填写并导入样本模板、启动分析。完成样本模板导入后,可批量运行任务,当客户在看到任务状态为 completed 时,代表任务已完成,即可查看报告部分(详见3.4部分)。
步骤一:上传数据
与使用场景一(手动投递)步骤一一致(详见3.2.1步骤一:上传数据)。
步骤二:构建参考基因组(可选)
与使用场景一(手动投递)步骤二一致(详见3.2.2步骤二:构建宿主参考基因组)。
步骤三:样本信息录入表格下载
- 点击导航栏【Data】,选择【Table】-【Download】(如图3.14), 点击【Workflow template】,选择MSAP模板下载

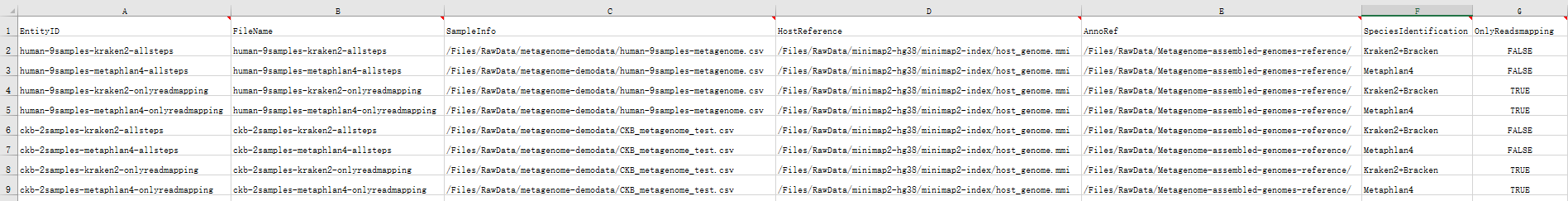
- 打开后MSAP 样本模板Excel如图3.15

步骤四:样本信息导入
- 该使用场景条件下,样本导入表格需填写工作表(图3.15)。该场景表示对已测序完成的样本数据在导入表格后,直接进入分析。
Excel注意事项
导入文件路径必须在云平台已存在,若不存在文件,则不会生成任务。
模板中所有参数均为必填项。导入数据中,必填项字段不得为空。
Excel中的EntityID需唯一。
Excel中不能合并单元格,单元格内容前后不能有空格或特殊字符。
分析样本录入(图3.16)
Excel 各字段意义
EntityID:为流程分析过程中关联任务和数据的标识ID,可以填写FileName等分析所需的真实ID,一个EntityID对应一个任务;
FileName:输出文件名前缀;
SampleInfo:包含样本名(SampleName)、样本测序文件(FQ1和FQ2)和分组信息(Group)的表格; 由用户在excel中填写,保存为csv格式,上传至数据管理;其中,样本名由英文字母、数字和下划线组成,相同的样本名视为同一个样本,所有FQ文件将会合并分析;样本测序文件路径要求是绝对路径而非相对路径,双端测序数据分别填写FQ1和FQ2,不支持单端测序数据;
DCS云平台绝对路径如下图:

鼠标悬浮于文件旁边的🔗符号,复制红框中文件路径,输入至表格中的FQ1或FQ2,分组建议存在生物学重复,由英文字母、数字和下划线组成;

HostReference:宿主的参考基因组索引文件,如在步骤二(3.2.2 构建宿主参考基因组)自主构建,选择Task ID文件夹内的minimap2-index目录下的host_genome.mmi文件;
AnnoRef:公共数据库中的Metagenome-assembled-genomes-reference文件夹(需要先从公共库Metagenome_assembled_genomes_reference复制到项目的数据管理),用于物种和功能注释;
SpeciesIdentification:用于物种鉴定的工具,仅支持MetaPhlan4或Kraken2,只能填写Kraken2+Bracken或者MetaPhlan4,默认使用Kraken2+Bracken。
OnlyReadsmapping:是否仅运行基于读长分析步骤,默认为false即运行基于读长分析和基于组装分析。
填写完毕后如图3.16:
- 配置好样本模板的分析样本录入工作表后,回到【Data】界面,点击【Table】-【+Add table】(图3.17)
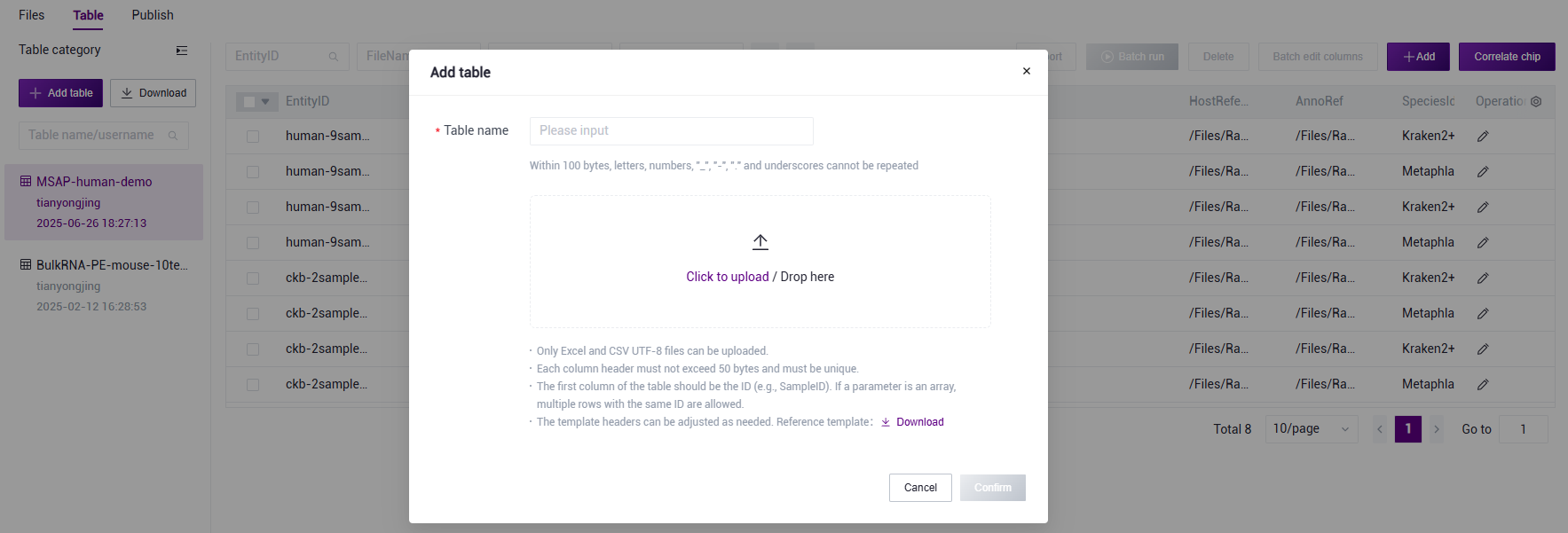
图3.17 样本信息导入步骤一
- 点击【Click to upload/ Drop here】浏览并选择已填好样本信息的表格,点击【confirm】(图3.18),上传完成后文件会在目标文件夹中显示

- 点击导航栏【Workflow】进入流程分析页面,在搜索框输入MSAP,点击【Run】(图3.19)
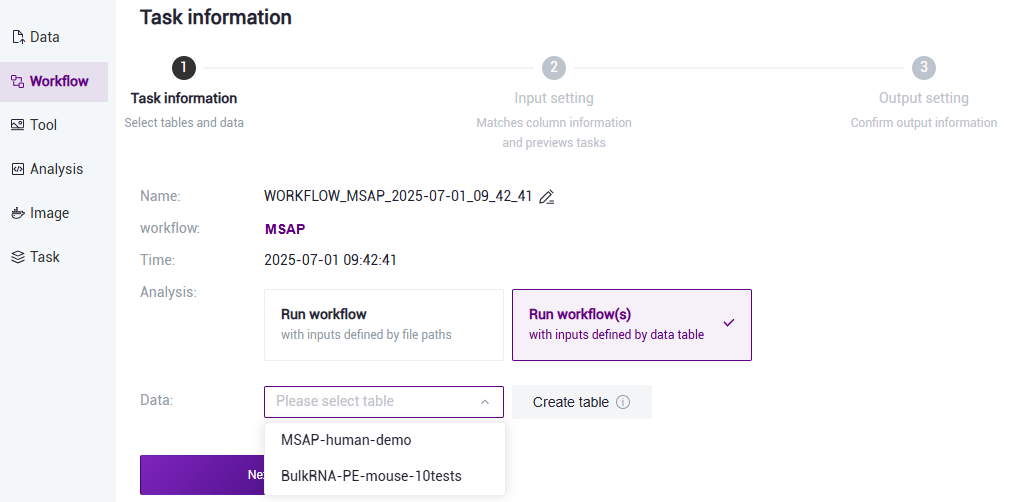
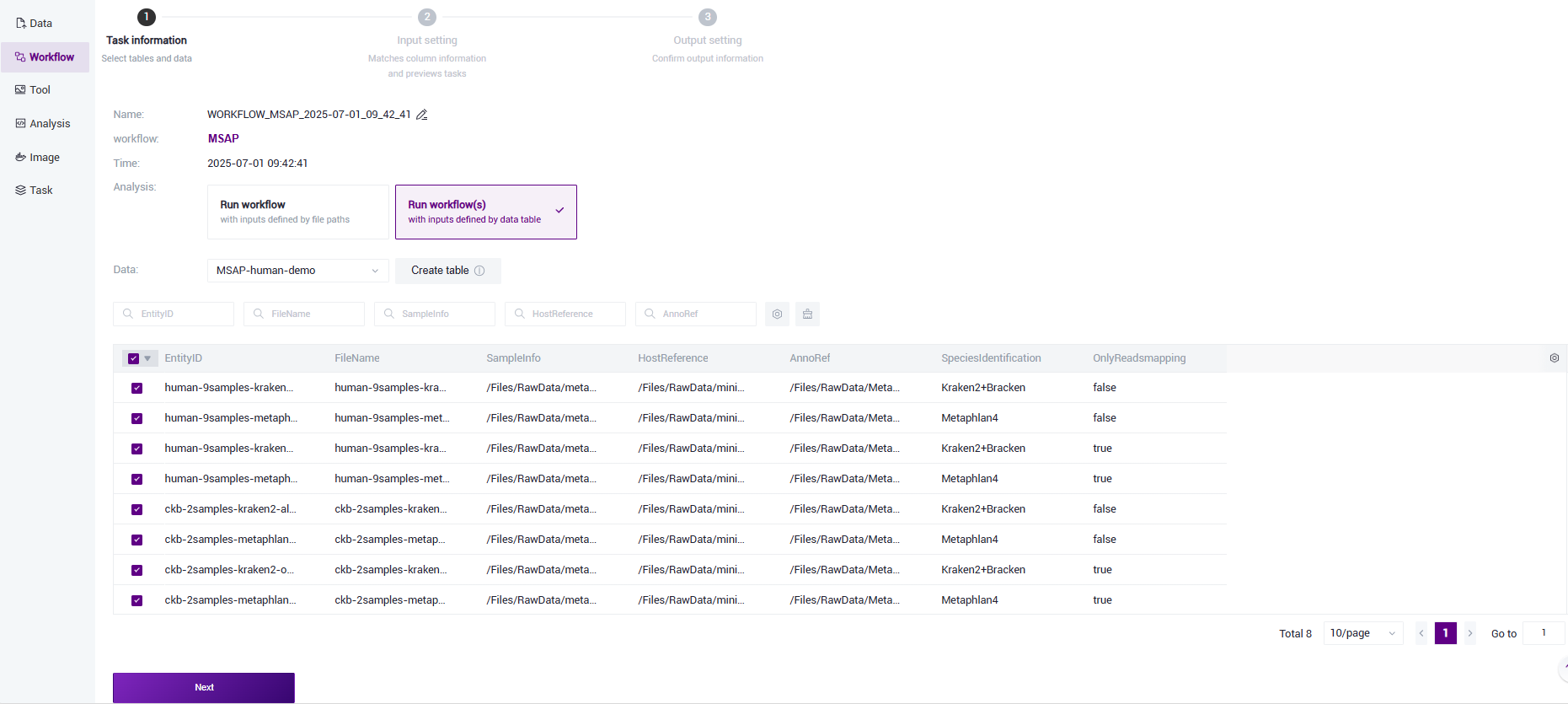
- 选择Run workflow(****s),点击 Please select table处,选择在本小节3)中导入的表格(图3.20),选中所需的行,点击【Next】(图3.21)
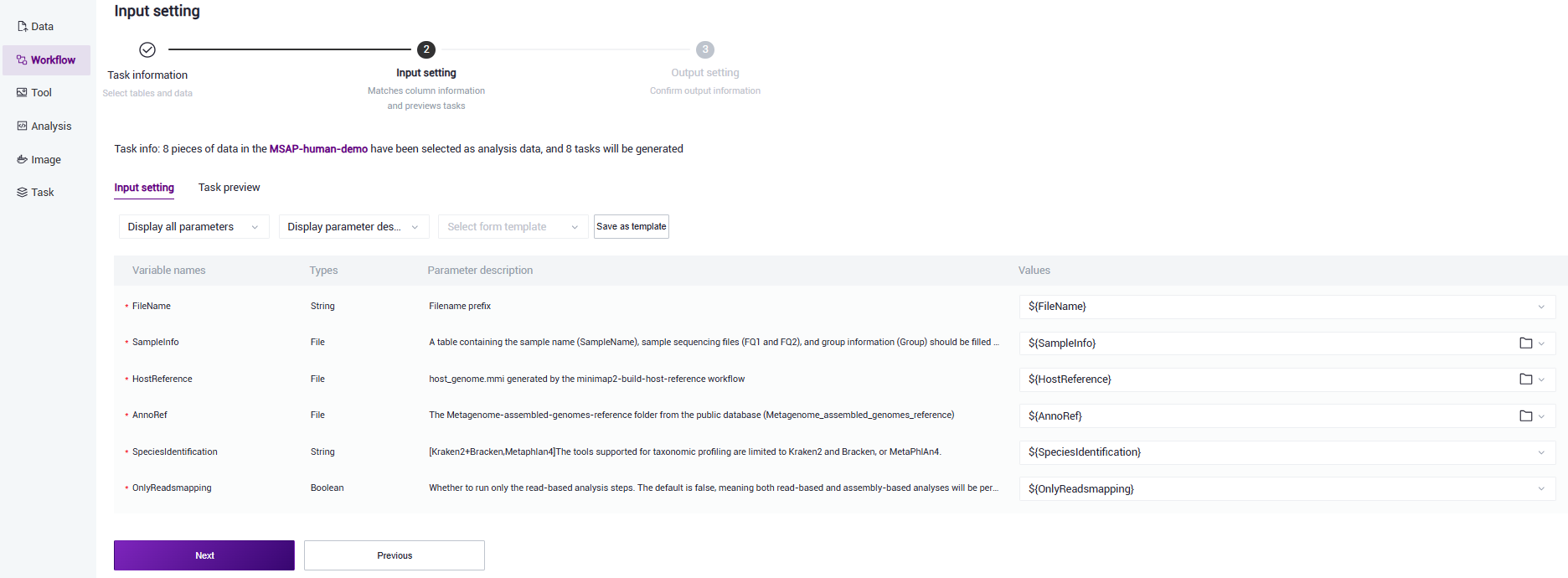
- Values一般会自动填充,仅需要检查填充是否正确,若需修改则在Values处点击
 并选择对应的值(如图3.22)
并选择对应的值(如图3.22)
步骤五:启动分析
点击【Run】启动分析(图3.22)
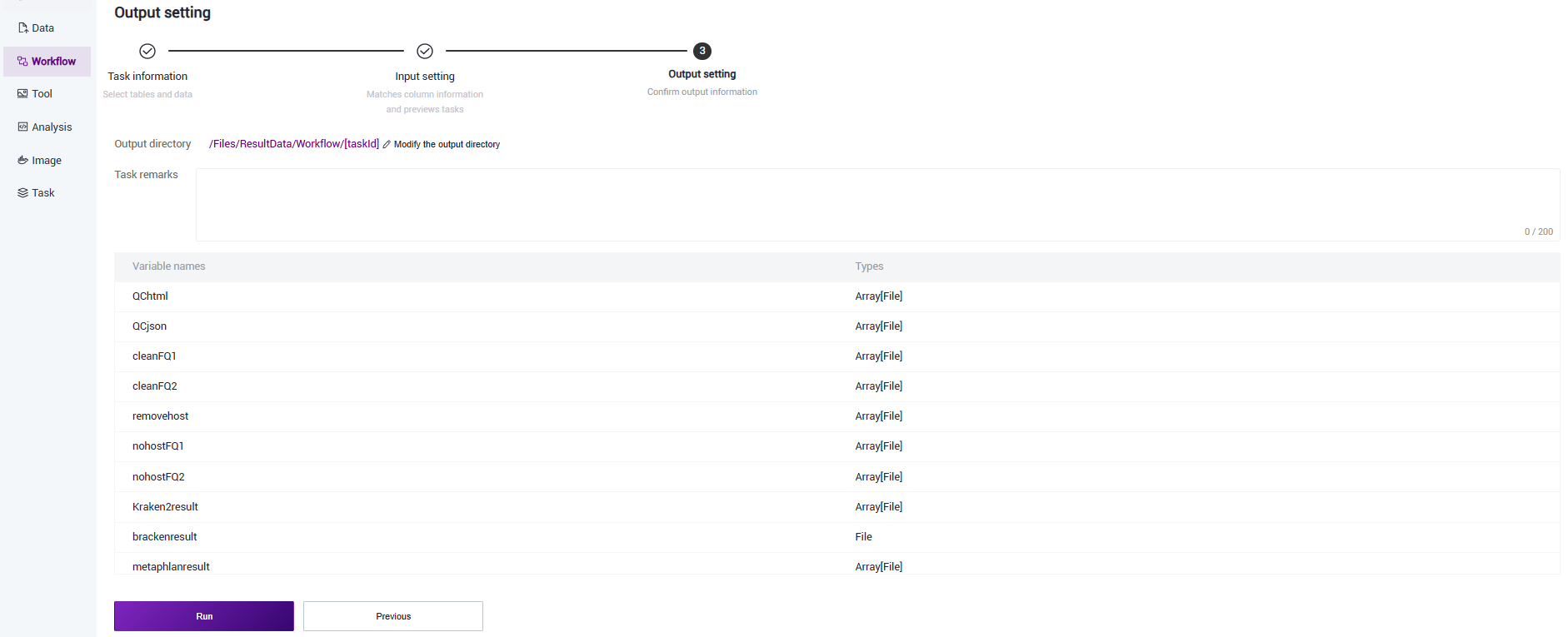
图3.22 启动分析
报告查看及结果文件下载
报告查看
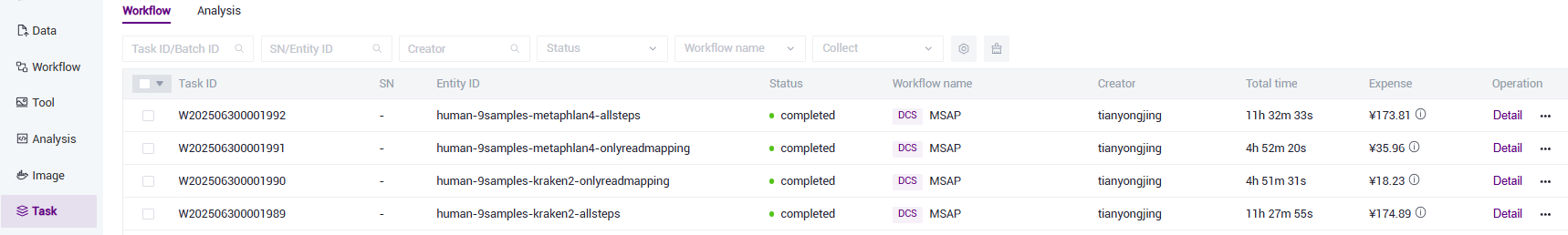
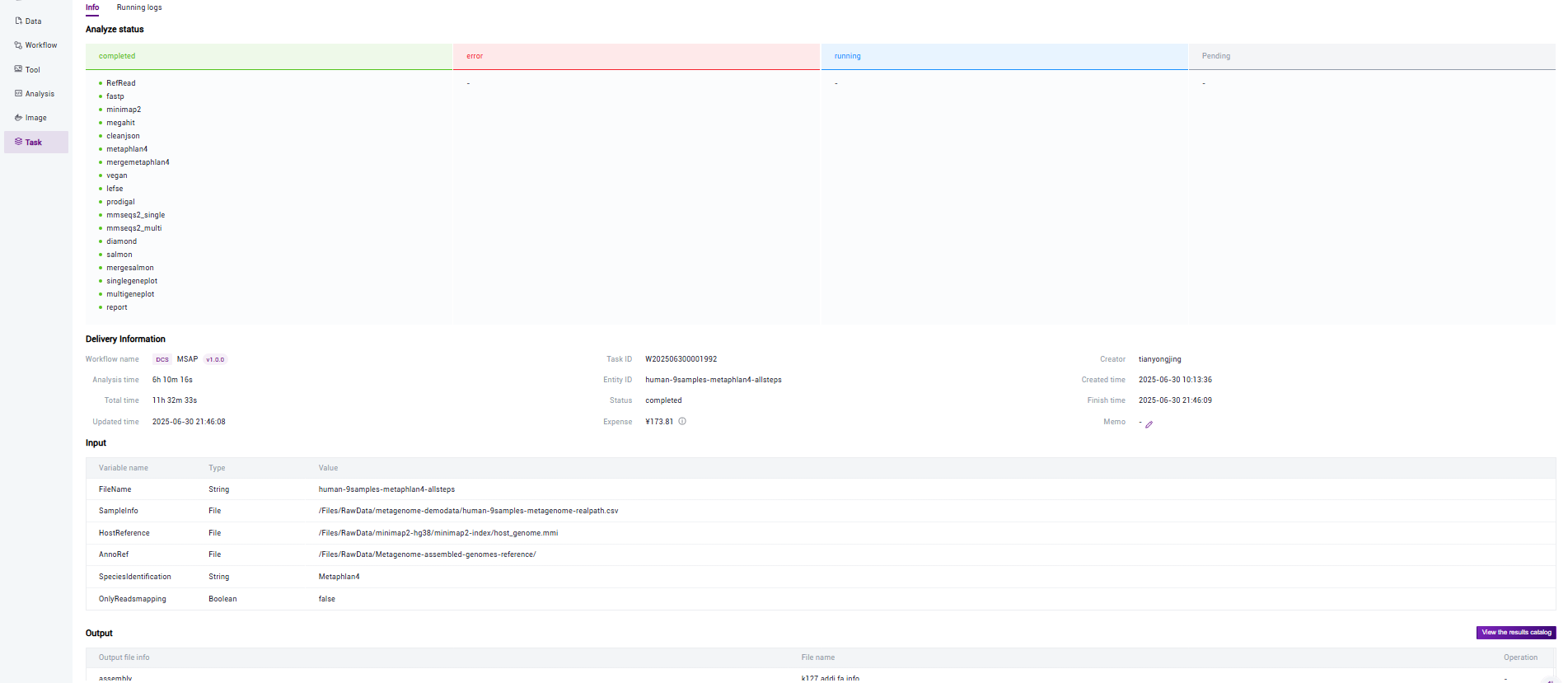
点击导航栏【Task】,当任务状态显示为![]() completed时,代表任务已完成,点击【Detail】即可查看结果部分(如图3.23),点击【View the results catalog】即可跳转到生成结果文件目录(如图3.24)
completed时,代表任务已完成,点击【Detail】即可查看结果部分(如图3.23),点击【View the results catalog】即可跳转到生成结果文件目录(如图3.24)
报告下载
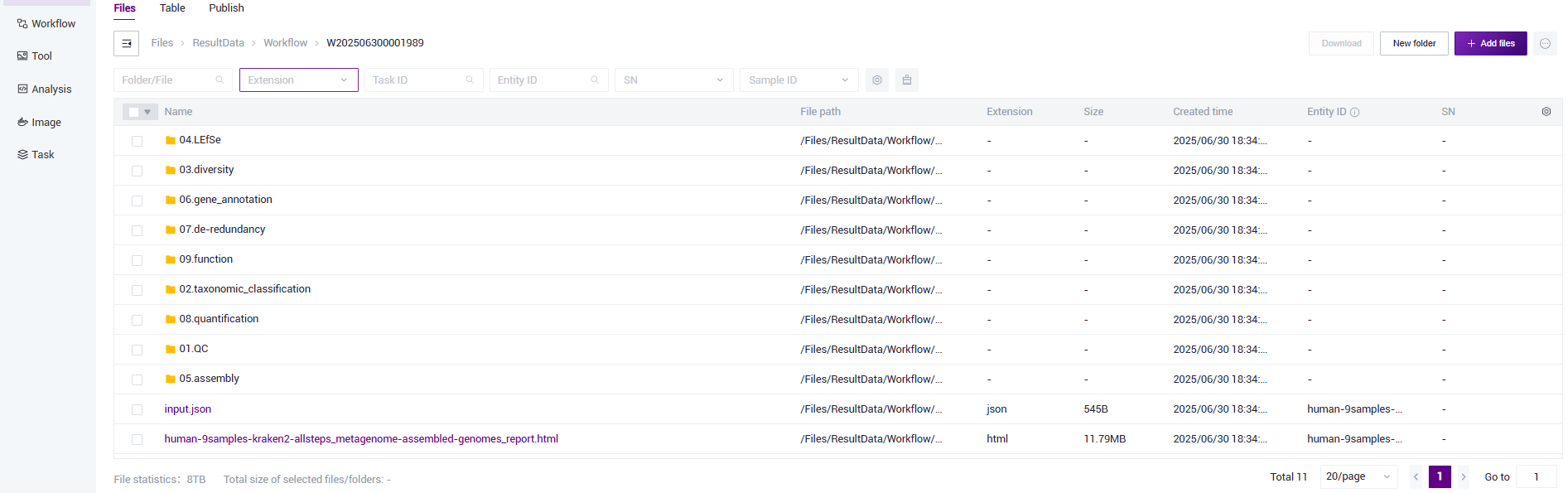
- 点击导航栏【Data】,进入数据管理页面,根据任务的Task ID进行搜索,点击进入Task ID文件夹(如图3.25)
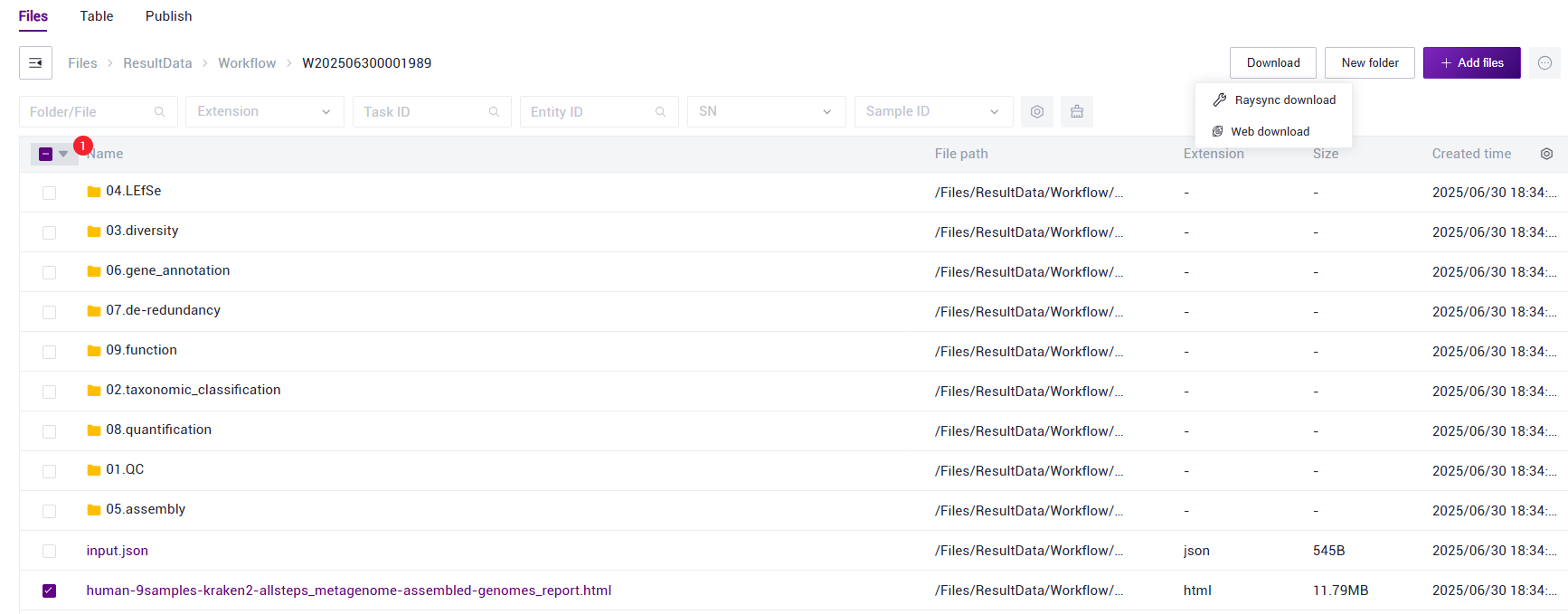
- 选中 ***report.html 文件,点击【Download】-【Web download】(如图3.26)
结果展示
MSAP样本报告展示