single_cell_RNA-seq
single_cell_RNA-seq
单细胞测序介绍
传统意义上在整个个体、器官或组织水平上使用组学方法进行的相关研究越来越丰富了我们对生物学的理解。虽然这些方法为研究生物学问题开辟了新天地,但由于在分析样品制备过程中通常以一群细胞为一个样本,分析结果仍然是所研究效应的均值。事实上,生物体中没有两个细胞是完全相同的,每种细胞类型都有不同的谱系和独特的功能,对组织和器官生物学产生影响,并最终定义机体整体的生物学功能。所以传统组学方法仍存在细胞异质性的困扰,而解决细胞异质性问题的最好方法就是单细胞分析技术。
一般单细胞测序流程如下图所示,包括细胞悬浮液制备、细胞分离、细胞标记、文库制备、测序及生物信息学数据分析等。下面我们重点来看单细胞的数据处理和分析方法。
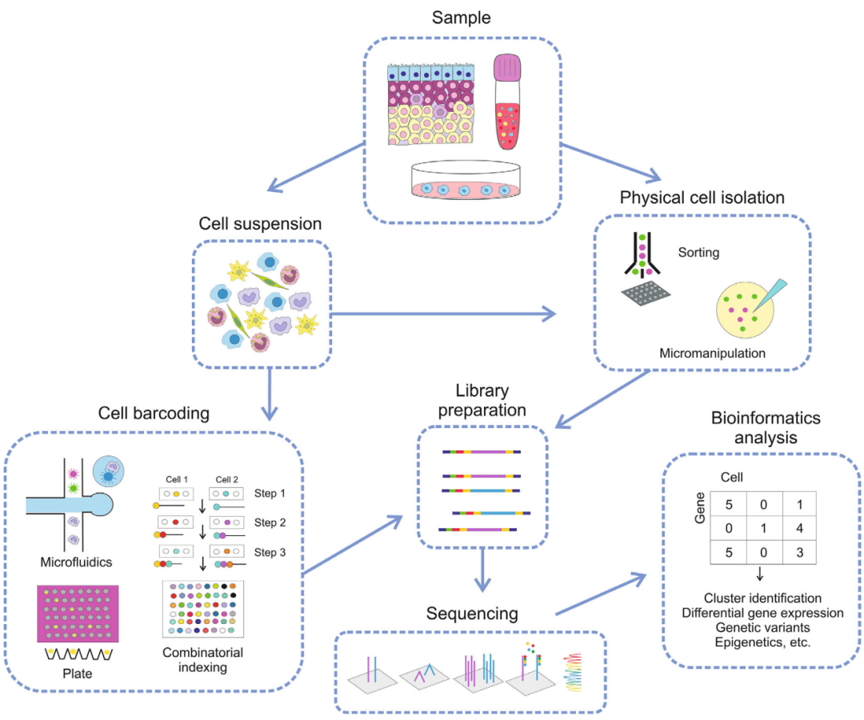
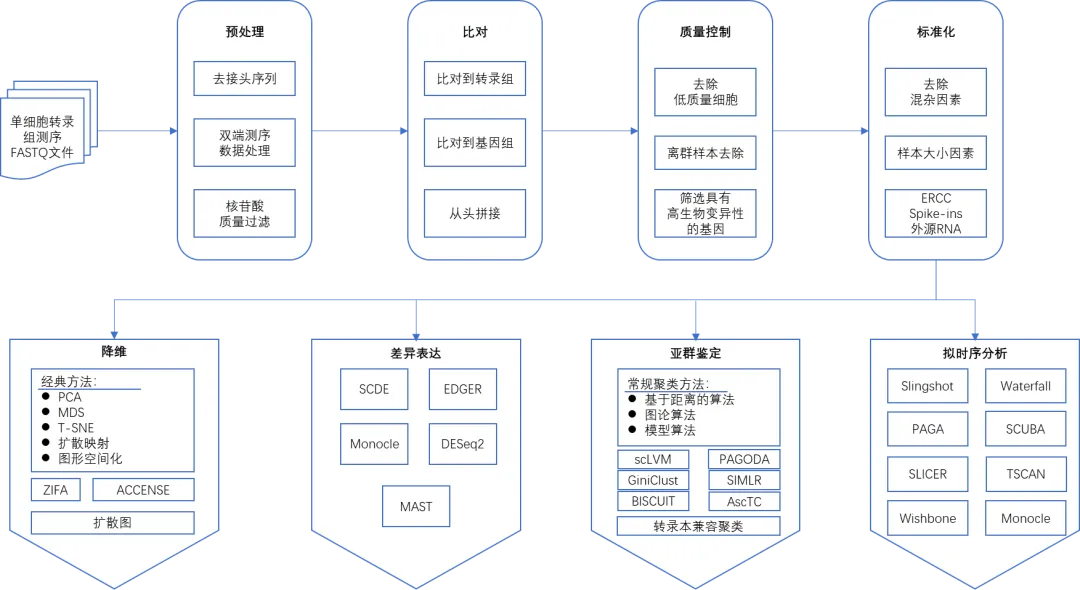
拿到单细胞测序数据之后,我们常做的分析有细胞周期评估、细胞聚类、识别差异表达基因、细胞轨迹推断和RNA速率分析、细胞通讯、基因变异信息、细胞系统发育和染色质可及性等,如下图所示,展示了各项分析内容及每种分析方法的工具选择。
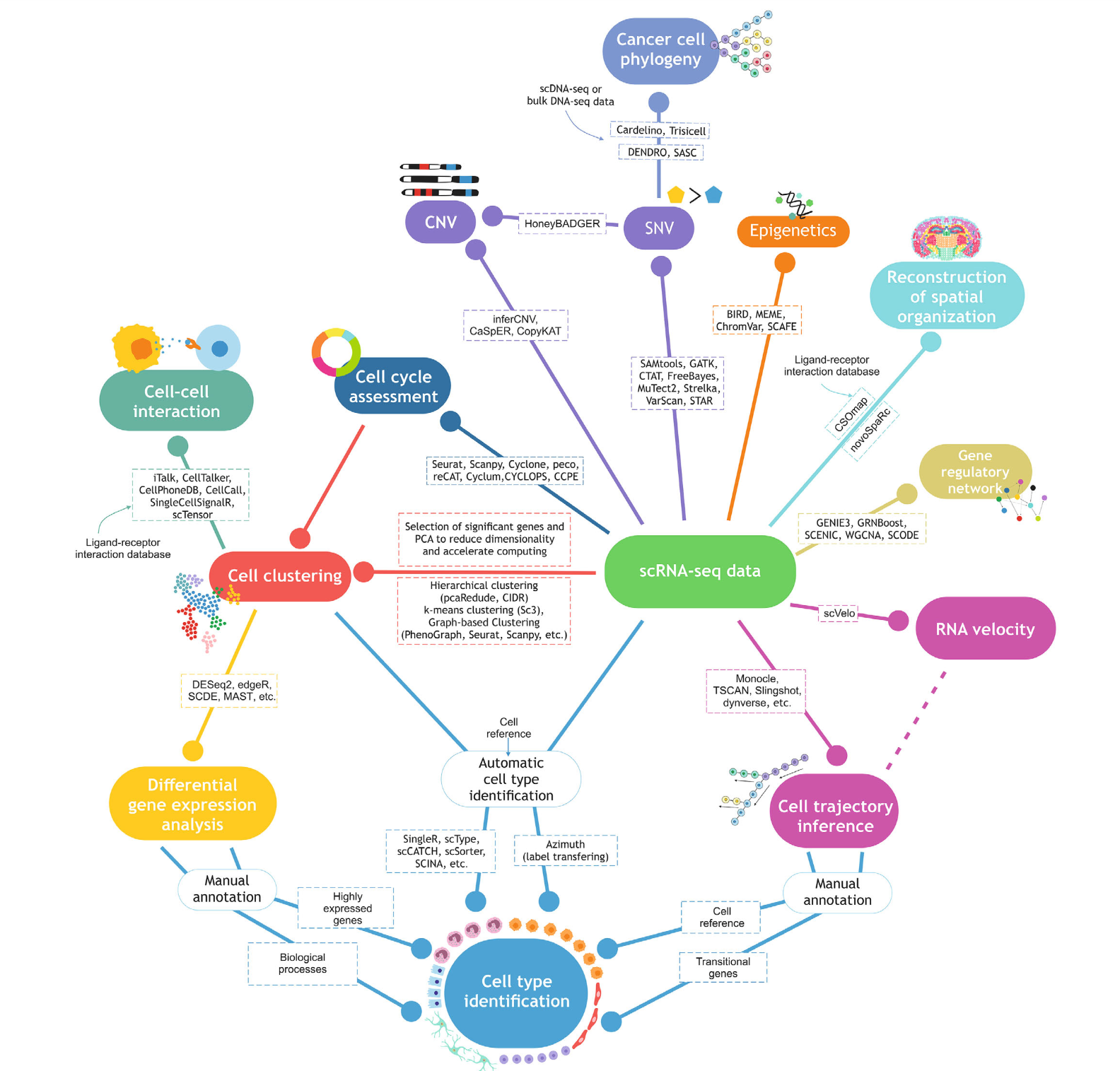
scRNA-seq除了用于确定细胞周期阶段、鉴定细胞簇、分析差异表达和信号通路以及细胞分型等,还可以用来研究CNV、SNV、染色质状态等各种遗传特征,以及组织和器官的空间信息。当然,单纯基于单细胞测序数据的话,一些分析方法还很受限,但随着生物信息学分析工具的快速发展、单细胞RNA/DNA文库制备方案的改进以及多组学分析平台的开发,单细胞技术将会有更大的应用空间。
基于DNBelab C4高通量单细胞RNA-Seq
DNBelab C4平台基于液滴微流控策略,可以实现高效细胞捕获,完成高效的mRNA反转录和扩增后,结合华大DNBSEQ™高通量测序平台和配套的单细胞数据分析软件,进行高性价比单细胞转录组文库测序和数据分析挖掘,可广泛应用于研究组织异质性、胚胎发育、克隆进化、疾病机理和免疫微环境等。
产品优势
稀有细胞高效捕:高效捕获稀有类型细胞,实现新突破;
多种样本无挑剔:除常规活细胞悬液外,珍稀液氮速冻样本可实现细胞核水平研究;
测序数据高保真:搭配DNBSEQ™测序系统,不怕单个细胞数据弄混;
自主分析不求人:云平台交互式单细胞数据挖掘系统支持;
服务模式一站达:流式分选服务服务助力目的细胞分选,一站式实现从组织到细胞数据获取及细胞群验证。
DNBelab C系列高通量单细胞RNA文库制备试剂盒套装V3.0
sc3' RNA-seq技术,基于磁珠引物尾部设计polyT碱基,捕获mRNA polyA特异性互补配对,并完成细胞的分离标记,制备多个细胞混合的文库,再进行高通量测序技术,获取mRNA的3端序列,能够揭示单个细胞的异质性和基因表达情况,在肿瘤、发育生物学、微生物学、神经科学等领域发挥重要作用正成为生命科学研究的焦点。
作为华大智造单细胞系列产品之一,DNBelab C系列高通量单细胞RNA文库制备试剂盒套装V3.0在原版本基础上完成了全新升级,包括液滴生成仪器TaiM 4,加样载片,液滴生成试剂,高通量文库制备试剂和开源的数据质控分析包来支持全流程的单细胞测序分析。
暂时无法在飞书文档外展示此内容
实验流程
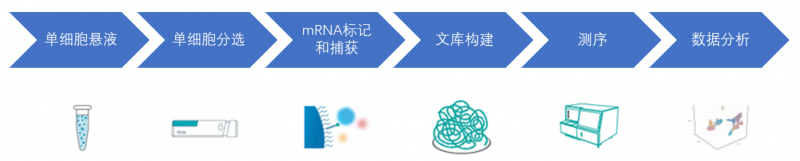
产品类型
低通量版本——预计产出4,000-5,000个单细胞/样本
标准通量版本——预计产出8,000-10,000个单细胞/样本
测序策略
推荐DNBSEQ™平台测序:
测序策略:PE100
测序深度:推荐平均50k reads/cell
测序数据量:2 lane/样本
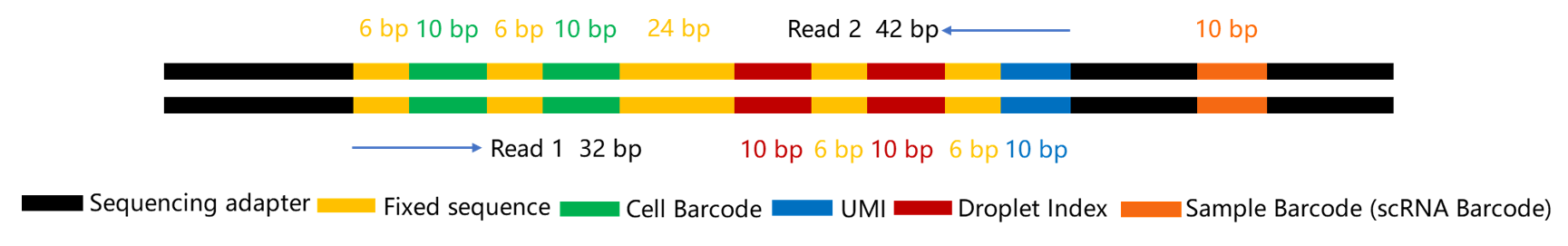
cDNA_library文库结构如下:
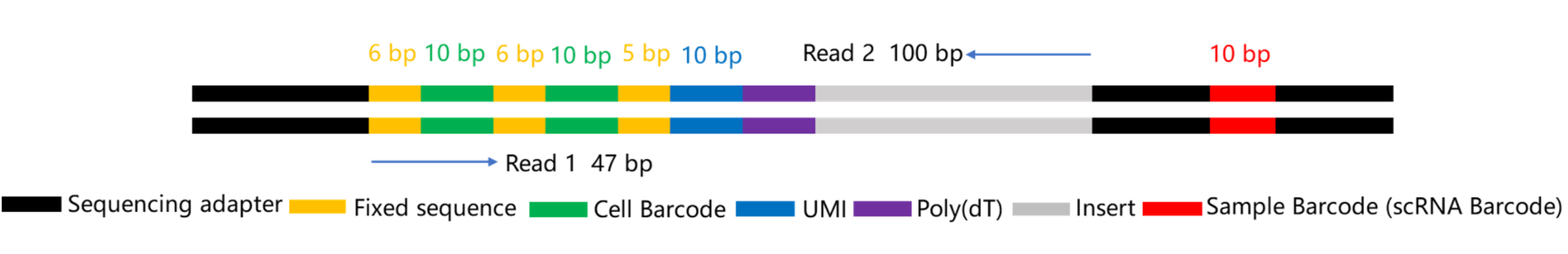
oligo_library文库结构如下:
更多产品信息请见:
https://www.mgitech.cn/Home/Products/new_bussiness_info/id/28.html
https://www.ebiotrade.com/ebiodesign/text/bgi/2022/0402/index.html
https://www.mgi-tech.com//Uploads/Temp/file/20240329/66066e0f968cd.pdf
生信分析内容
- 标准分析流程:
- 一般信息分析内容见下图:
云平台数据分析
基础分析
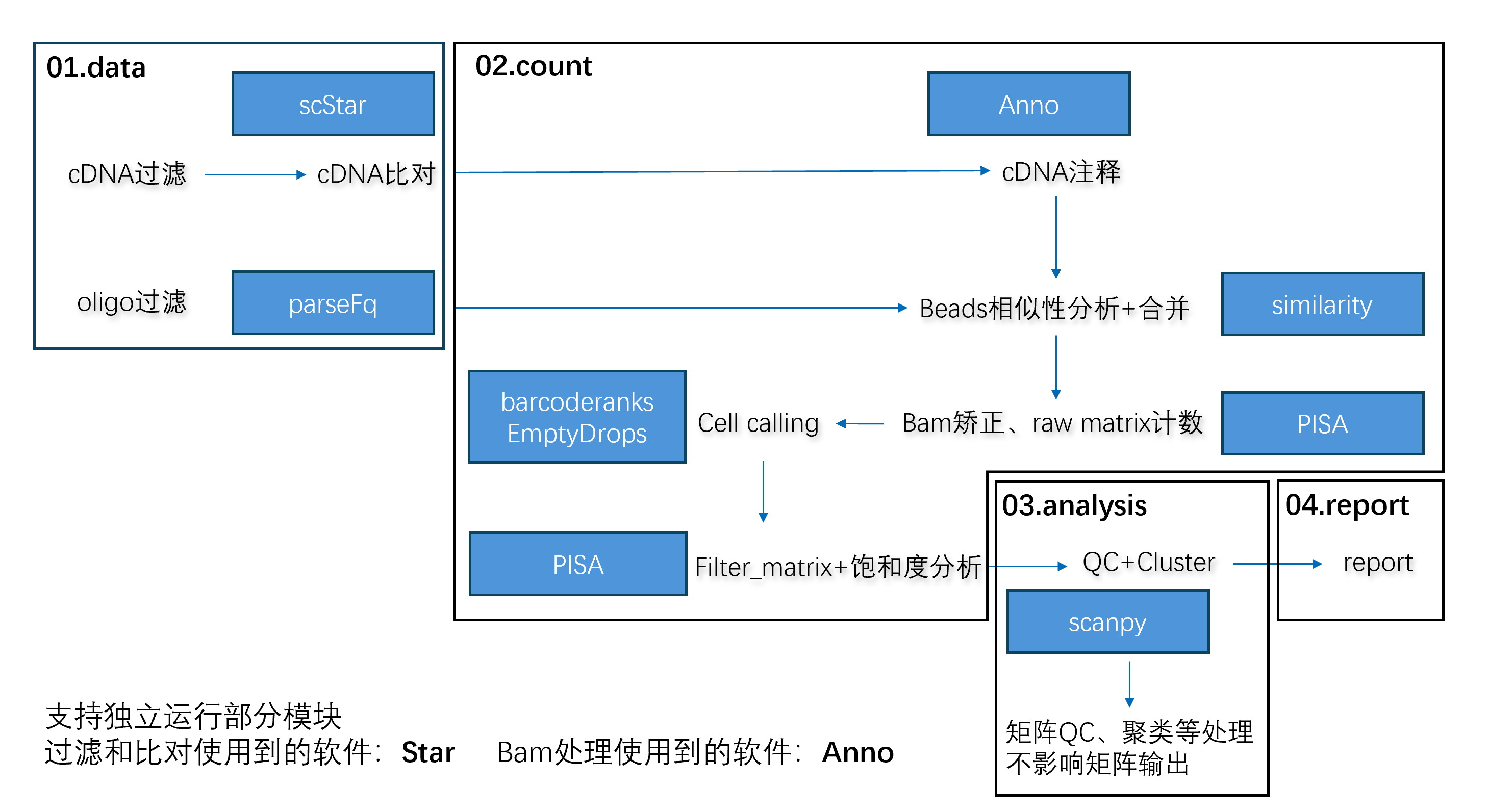
云平台已将github标准分析流程转为线上WDL分析流程(名称:scRNA-seq_v3.1.5),用户可以在网页上进行交互式分析,基础分析主要包含以下步骤:
RefRead:检查将用于reads比对和表达定量的参考基因组文件,包括包含参考基因组索引的公共目录、参考基因组gtf文件等。
M280parseFastq:解析细胞条形码umi和并对数据进行质控。
star:使用STAR软件(已做定制化修改)来比对参考基因组。
anno:根据gtf文件注释reads以对齐基因组区域,umis校正并生成原始计数矩阵。
cleanAfteranno:删除多余文件以节省存储空间。
getM280UMI:这一步包括beads calling 及合并。 在 scRNA-seq 中,beads通常用于捕获和分离单个细胞。 识别和过滤空滴和双胞,计算相似性并合并具有高相似性的beads,并对bam 文件添加cell标签。
statSaturation:根据之前步骤生成的采样数据文件,计算组织覆盖区域的测序饱和度。
count:计算合并bead后细胞的基因表达量,对细胞和基因进行统计,输出三个标准矩阵文件供下游分析。
splice_matrix:如果选择注释内含子,则会有一个剪接RNA矩阵和一个RNA速度矩阵。 这两个矩阵文件可用于深入分析细胞发育轨迹和方向。
QC:计算细胞过滤后的质控信息,包括基因数、总umis数、线粒体百分比。
Cluster:单个细胞的降维、聚类及注释信息显示
报告: 生成 HTML Web 报告,整合每个步骤的分析结果,并显示关键统计指标、测序饱和度图、聚类分析和注释分析结果。
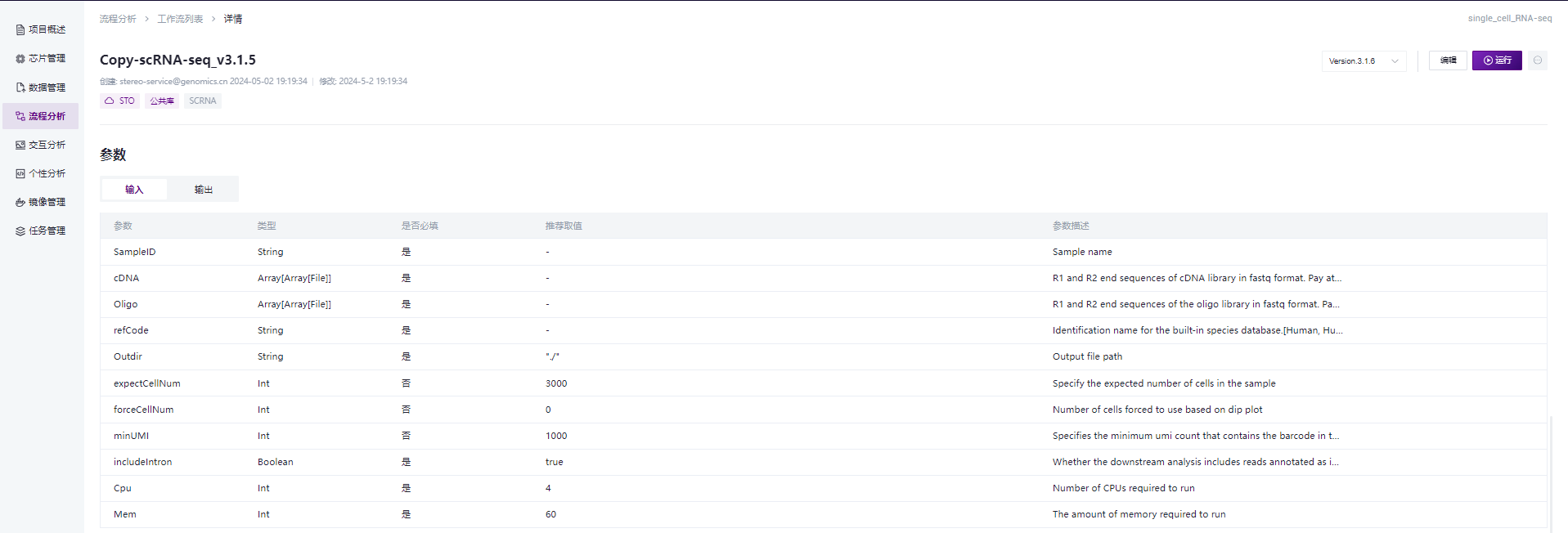
WDL流程输入参数及含义如下图所示:
具体操作步骤包括以下几步:
1. 从公共库复制流程,进入项目-流程分析-添加工作流,搜索scRNA-seq_v3.1.5,并复制到项目下;
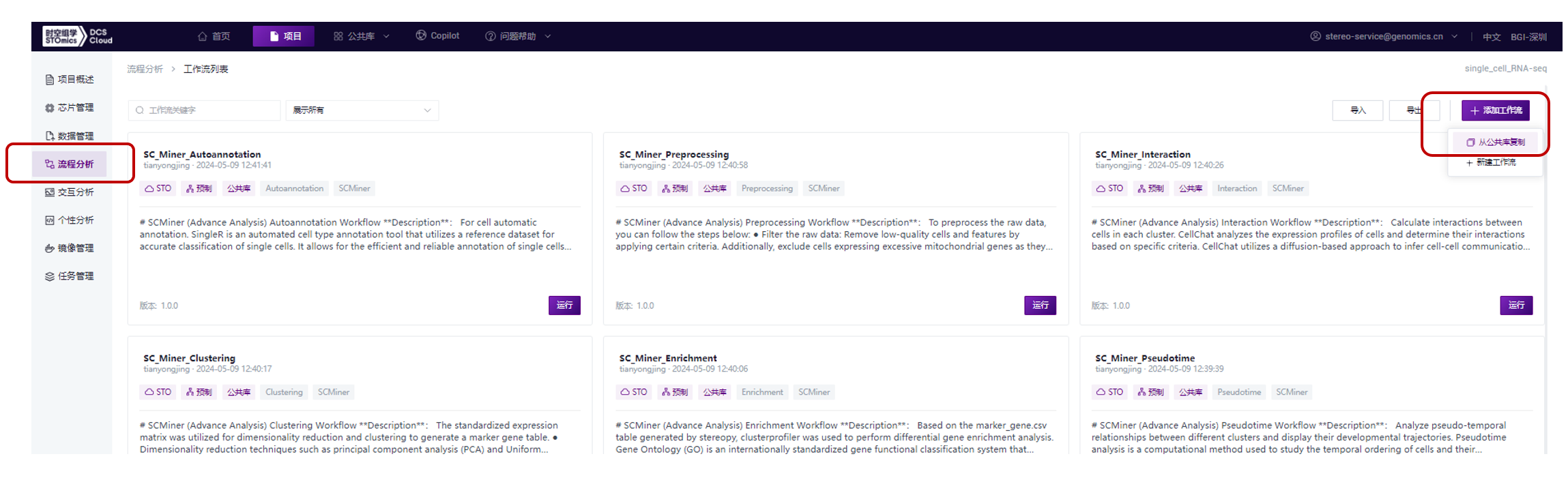
2. 运行工作流,找到上述从公共库复制过来的分析流程,点击“运行”按钮,如下图所示:
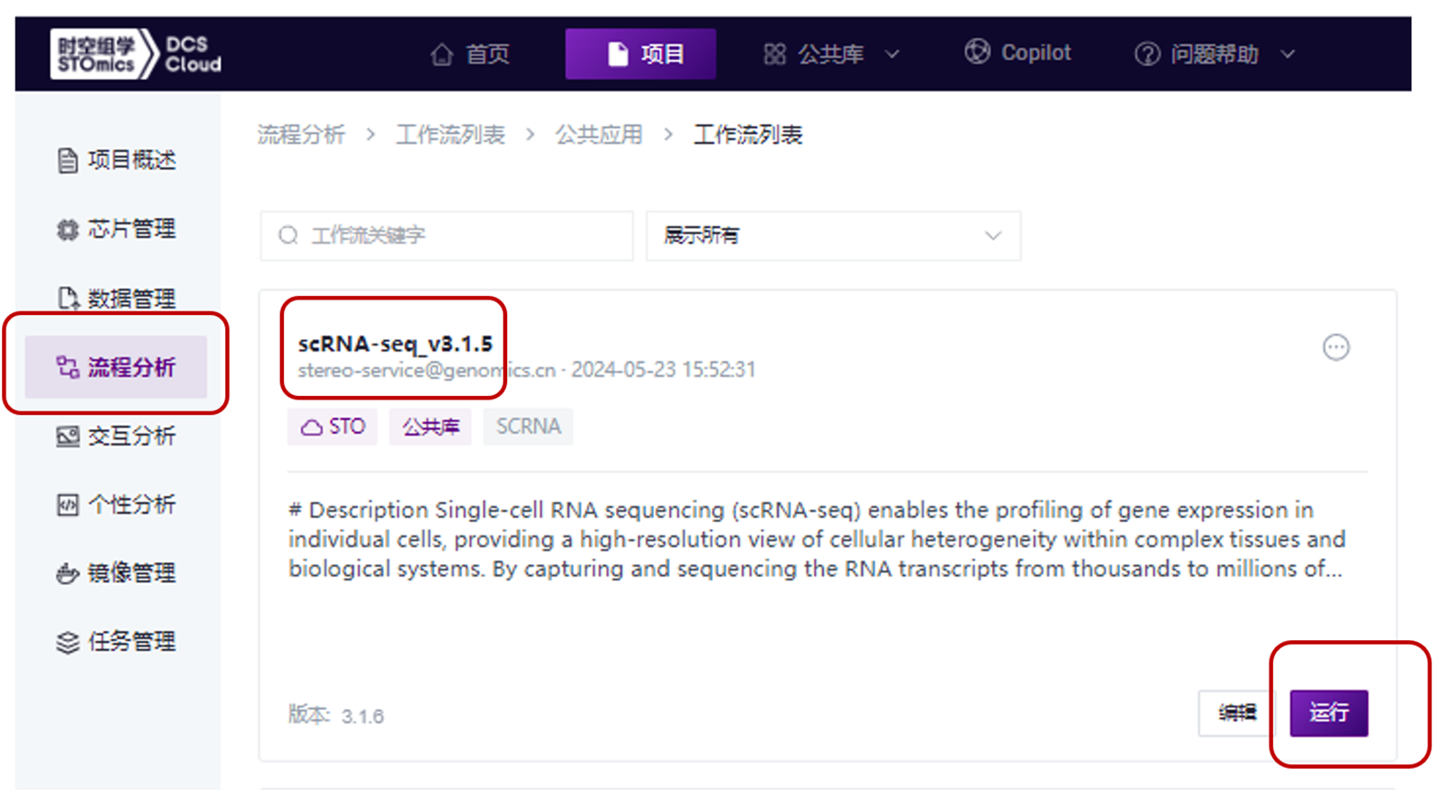
选择“运行界面输入”,填写实体ID信息,并点击“下一步”按钮,如下图所示:
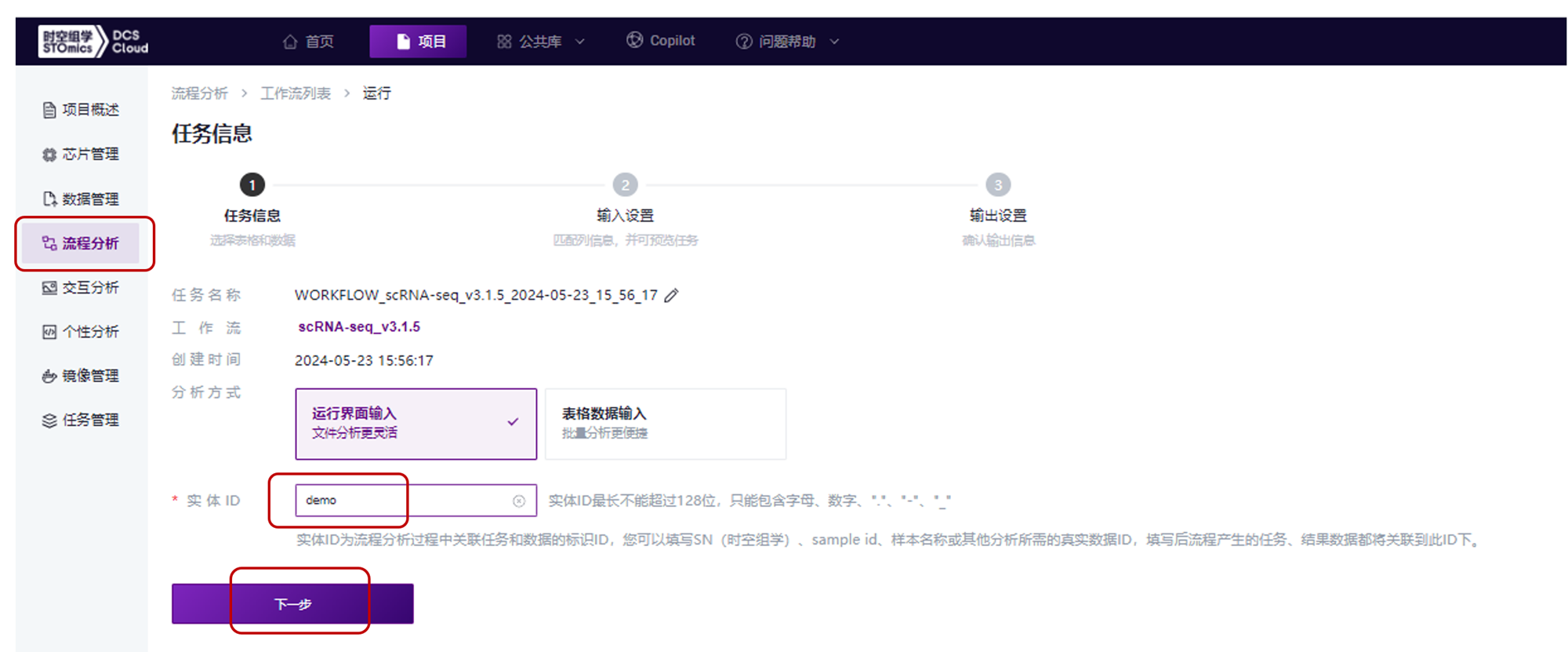
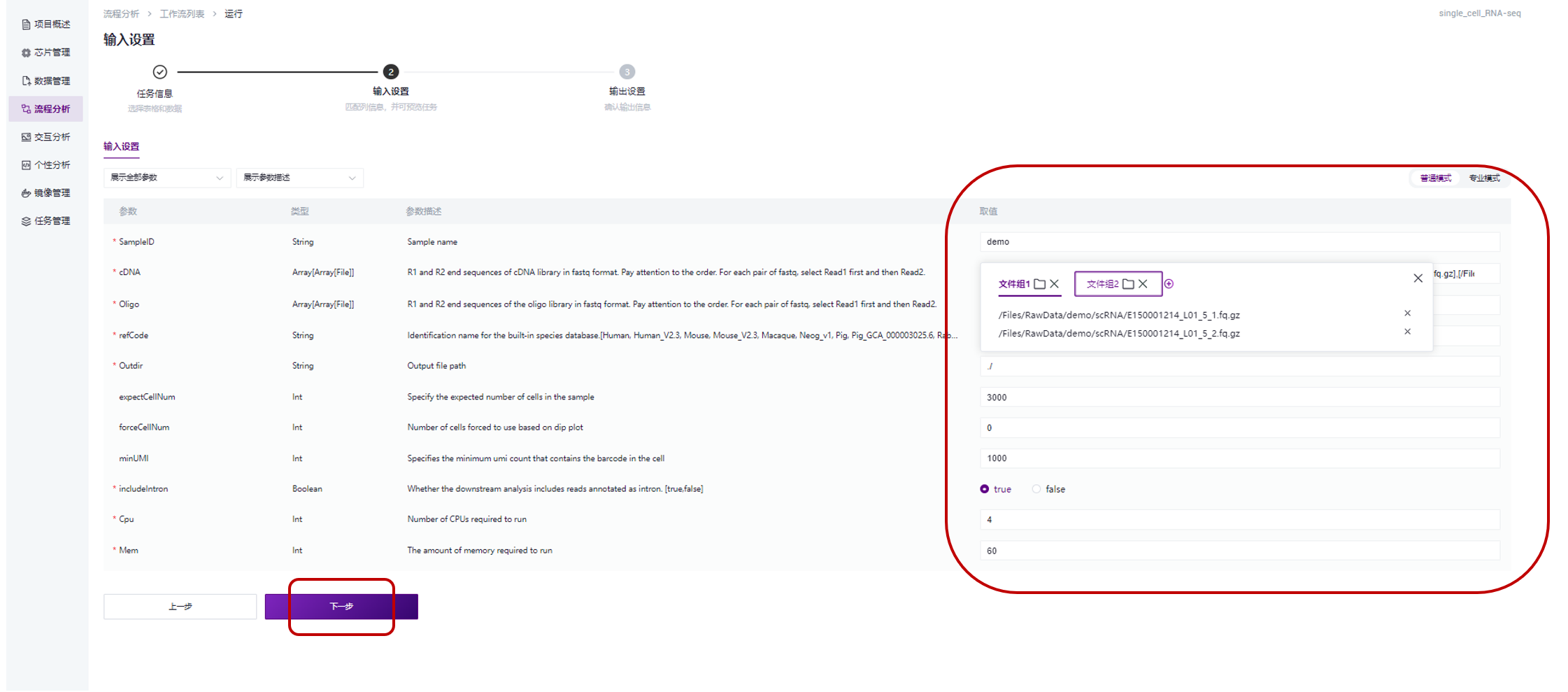
在参数输入设置页面根据提示(参数,类型及参数描述)选择对应的文件及参数,如下图(具体信息请参考后续任务详情-输入信息),并点击“下一步”按钮:
在上述跳转的窗口中修改输出目录和任务备注信息,点击“运行”按钮,如下图所示:
在点击运行后跳转的窗口,为前往任务管理与否的选择弹框,点击“确定/取消”按钮,如下图所示:
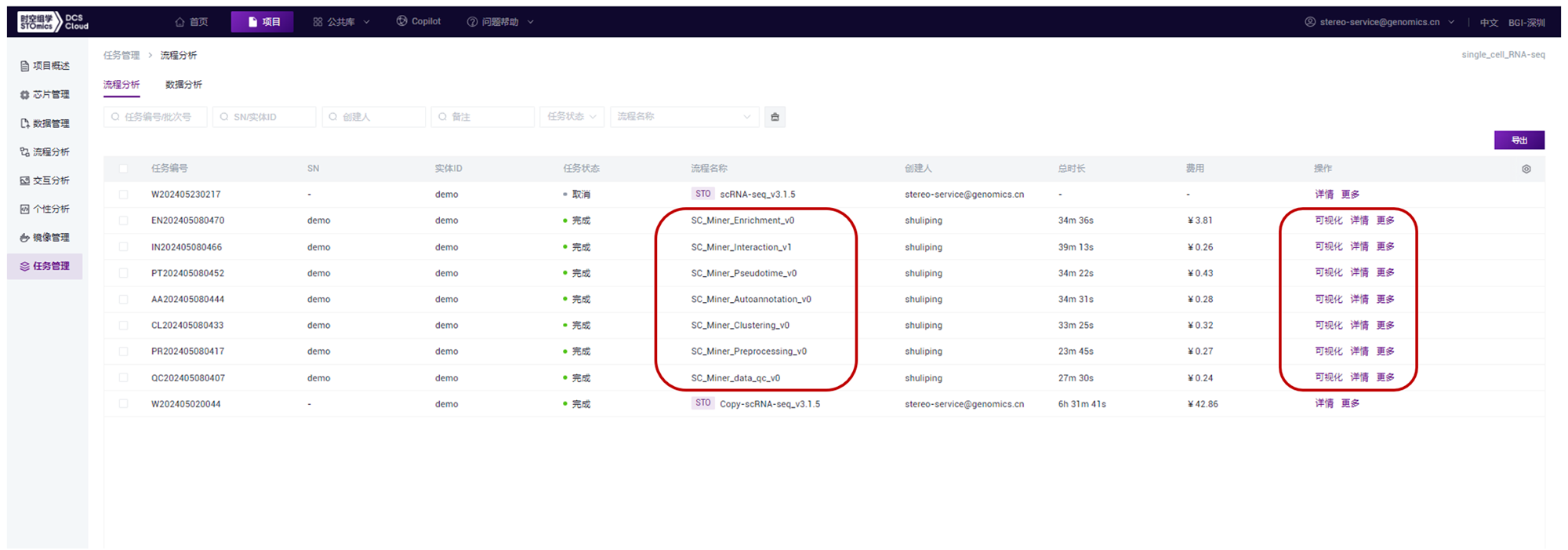
点击“确定”后,会跳转至任务管理,可以进行任务状态查看以及更多操作,如下图:
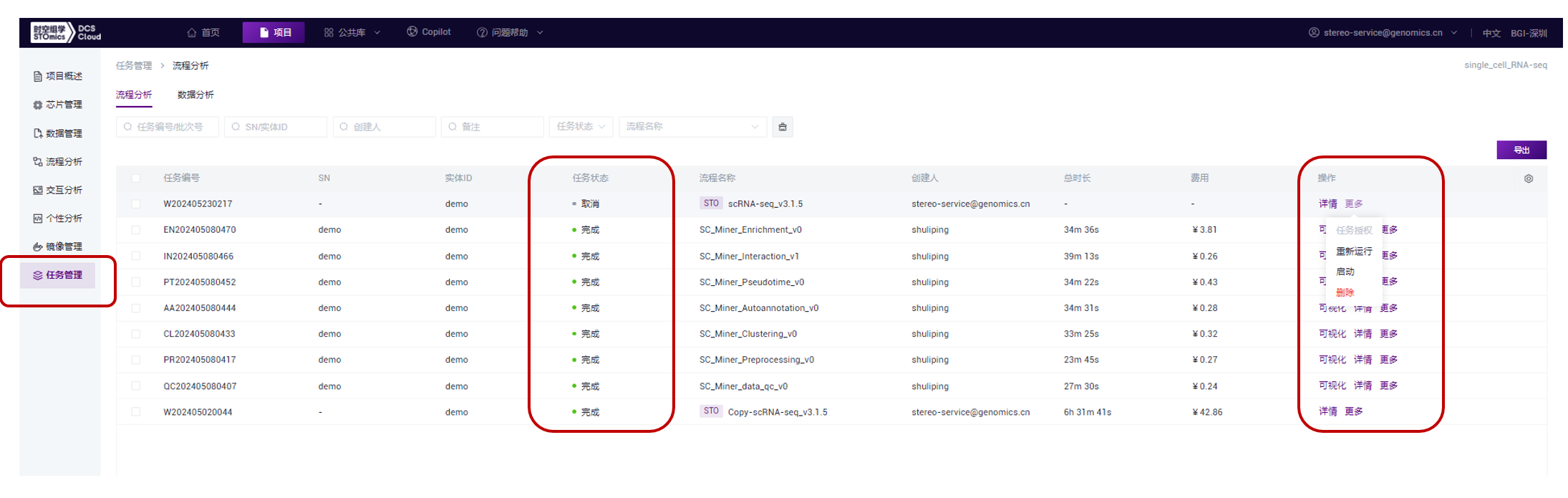
点击上述页面操作栏,“详情”按钮,可以查看任务详细信息,如下图,有任务分析状态、任务投递信息以及输入、输出等信息。
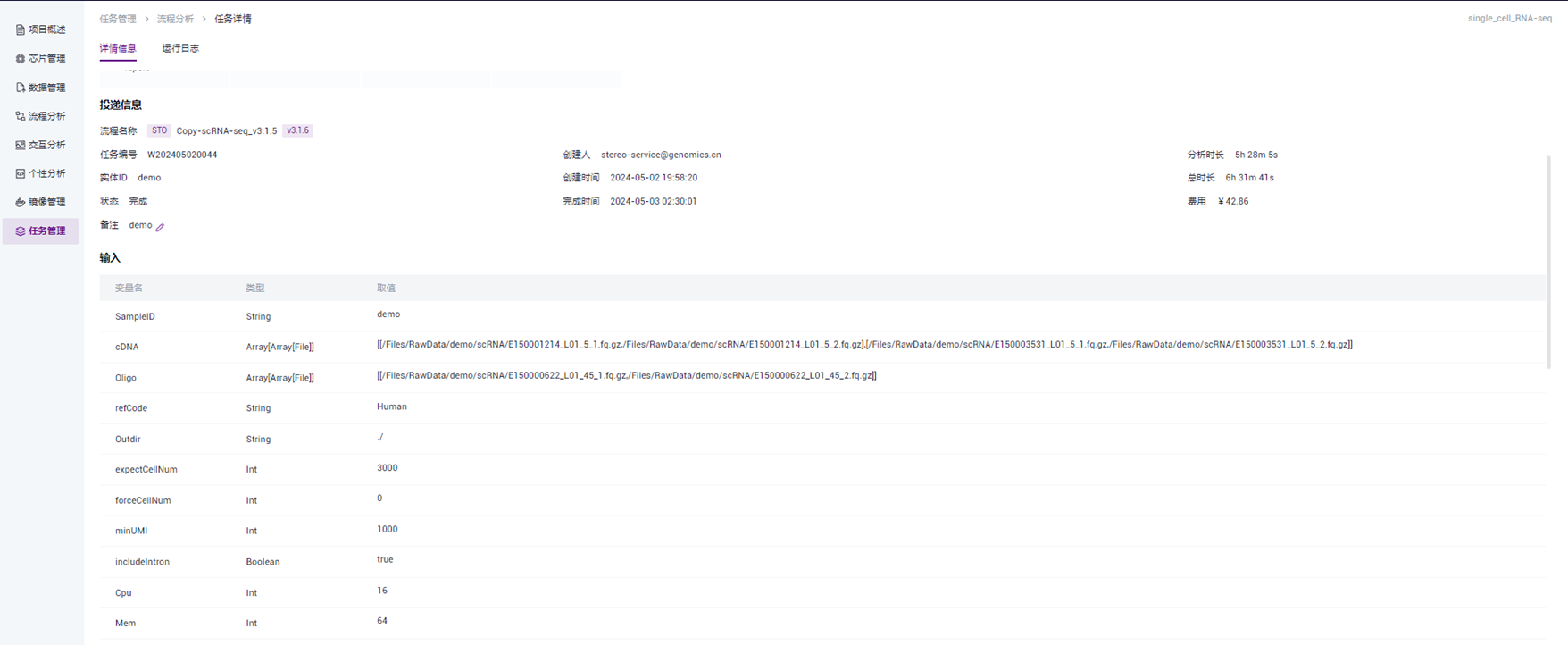
3. 查看结果文件,如下图,点击任务管理-详情页面,待任务完成后,下拉可见“查看结果目录”按钮,可以点击进去,根据需要,进行结果文件查看及下载等操作。
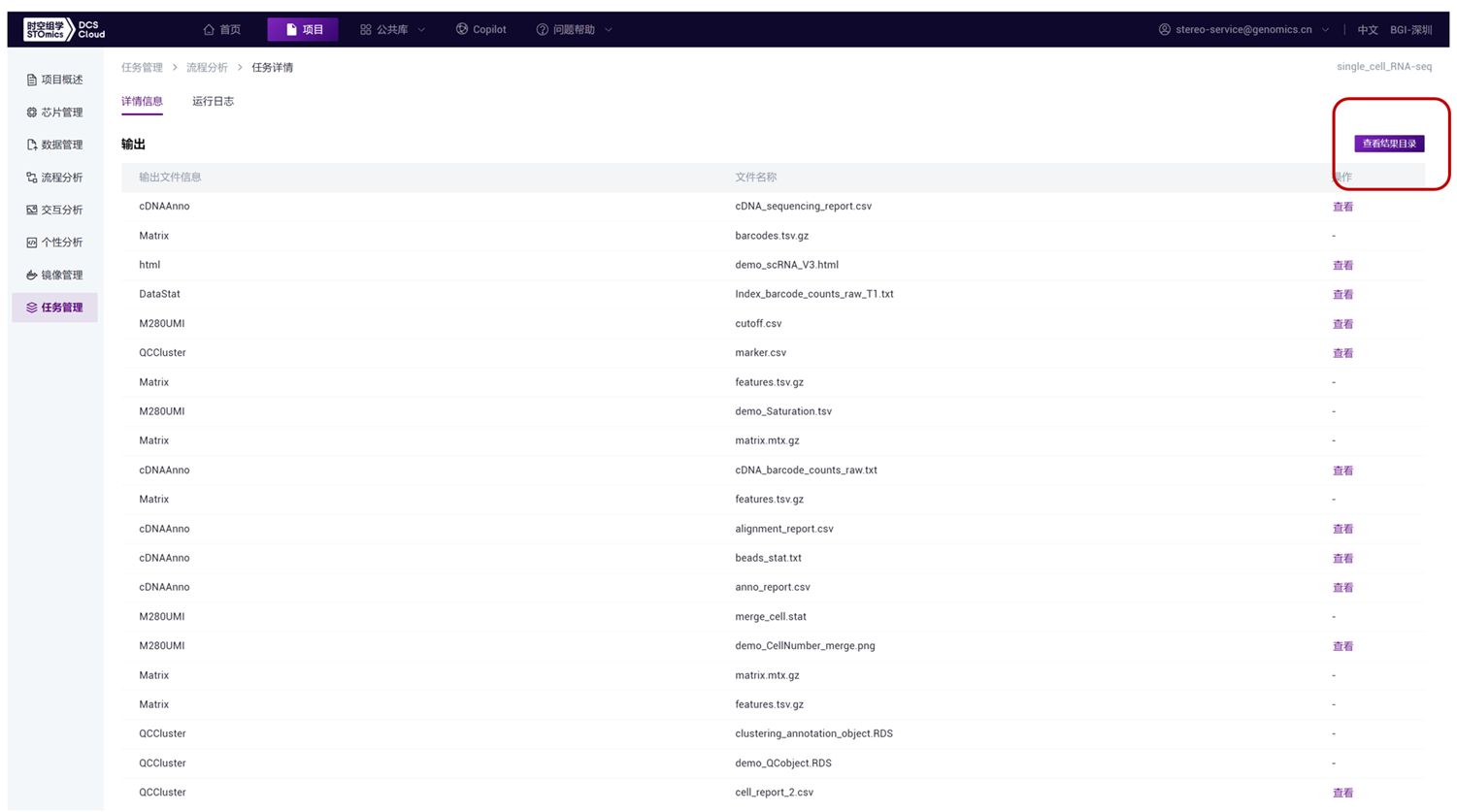
高级分析
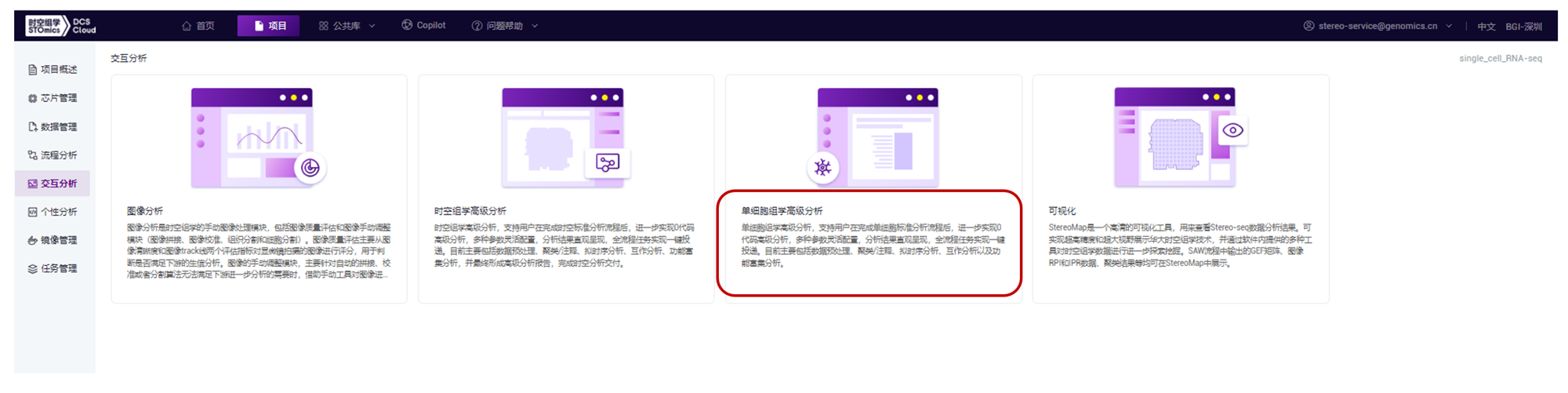
云平台开发了一套自动化、交互式单细胞高级分析流程,自动化完成基础分析后,进入交互分析-时空组学高级分析模块继续进行高级分析,如下图所示,该模块分析内容包括:数据预处理、聚类及注释、拟时序分析、互作分析、功能富集分析、分析报告生成7个步骤。
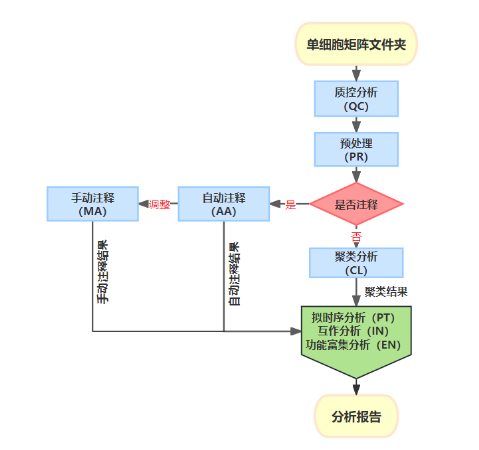
分析流程如下图:
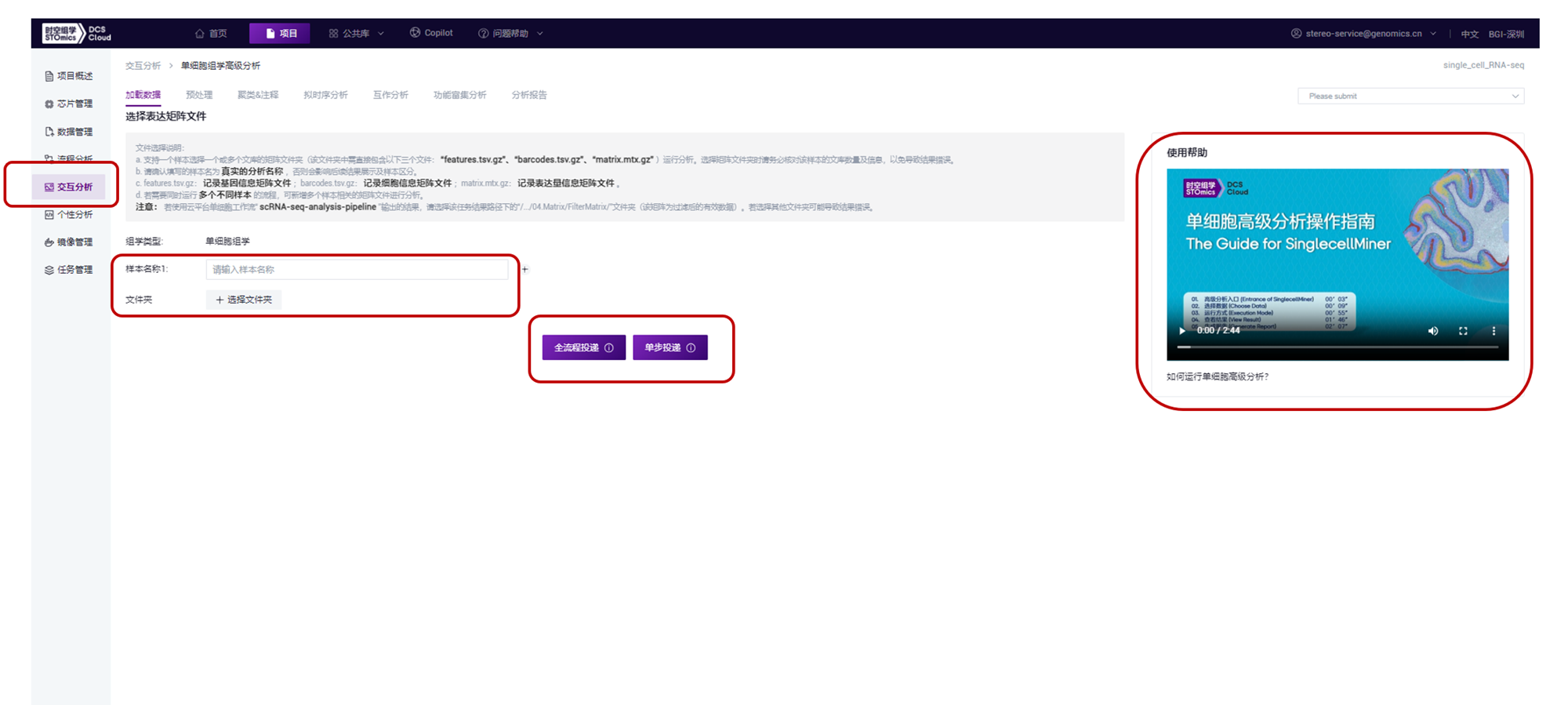
根据下图所示页面上提示进行样本名称信息填写、文件夹选择,根据实际情况,进行任务的全流程投递或单步投递;如果还有不确定的信息,可以点击页面上右边展示的使用帮助视频-“单细胞高级分析操作指南”内容,参考操作指南进行分析。
查看分析结果,如下图,可以查看高级分析各个主要步骤的分析结果,点击操作栏“可视化”按钮,进行结果在线查看及参数重置后重新提交任务分析,不断优化结果。
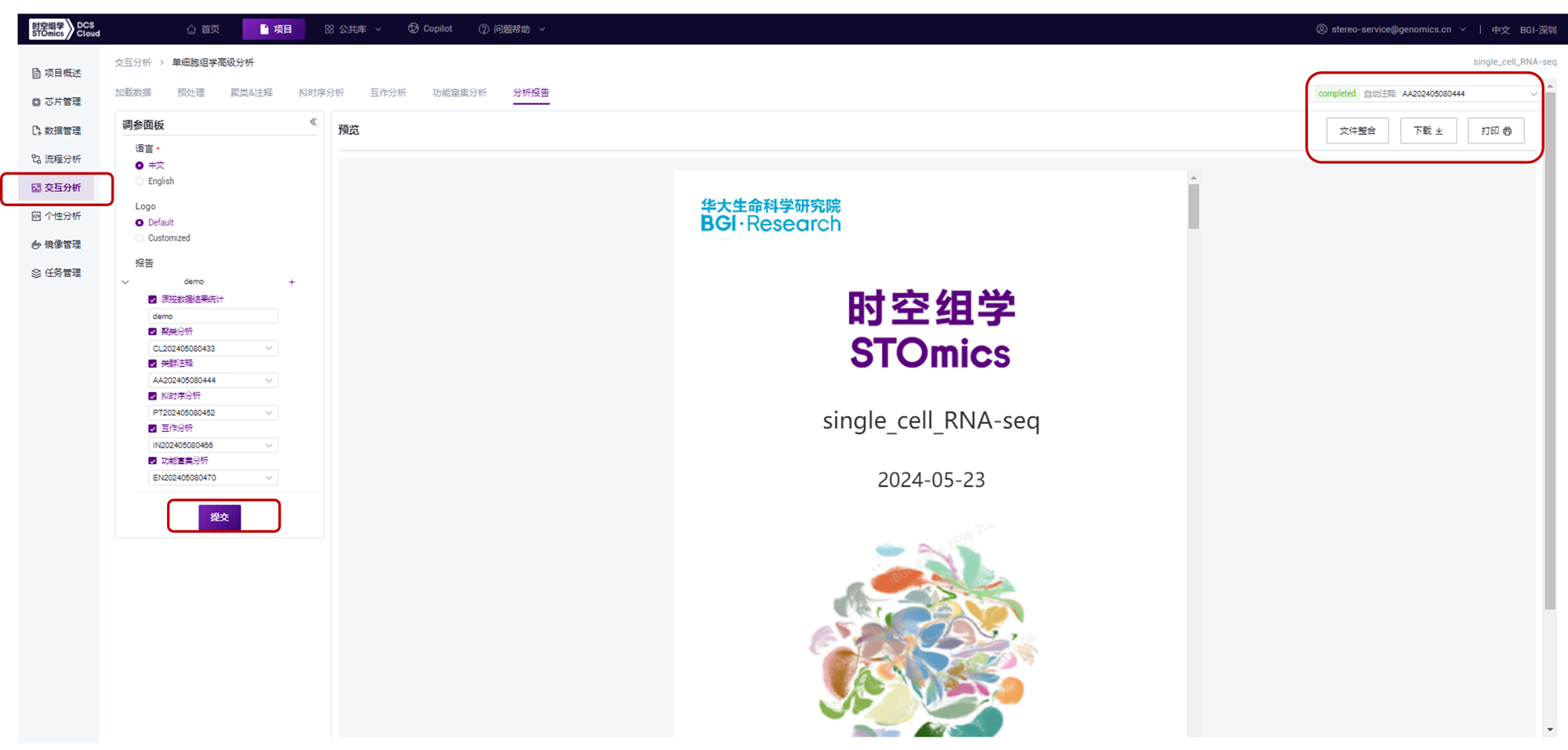
生成高级分析的分析报告:如下图,在完成各项高级分析后,在左侧调参面板下报告模块中选择想要生成报告的任务结果,点击“提交”按钮,之后可以生成报告,根据需要进行文件整合、下载和打印等操作:
高级分析详细使用说明请参考:
https://www.stomics.tech/helpcenter/zh/usermanual/E.Project/F.Tool/B.StereoMiner.html
更多单细胞数据分析方法介绍可参考:
https://github.com/MGI-tech-bioinformatics/DNBelab_C_Series_HT_scRNA-analysis-software
https://github.com/seandavi/awesome-single-cell
https://github.com/hbctraining/scRNA-seq_online
常见问题
Q1. 通过DNBelab C4平台进行单细胞RNA-Seq的组织样品,是否可以提供前期处理的方法?
答: 目前单细胞送样建议手册上有提供通用的组织消化方法,可供参考。
需要注意,不同组织存在组织及细胞的特异性,使用通用组织消化方法的效率会因组织类型差异,存在消化效率的差异。因此,建议客户自行使用组织特异性的针对性消化方法进行消化。
Q2. DNBelab C4平台分离的单细胞会有细胞大小的限制吗?
答: 细胞大小会有一定的限制,但限制条件相对固体芯片没有那么严格。同时,生产实验过程中,为了避免细胞出现结团情况,在进行细胞分离处理前,会做一个粗略的过筛处理。建议细胞大小<40um。
Q3. DNBelab C4平台单细胞RNA-Seq可以进行可变剪切等结构变化分析吗?
答: DNBelab C4平台单细胞RNA-Seq主要定位为基因表达定量分析为主,数据不建议进行可变剪切等基因结构变化分析。如需研究结构变异信息,可进行单管单细胞RNA测序。
Q4. DNBelab C4平台单细胞RNA-Seq采用的3’端扩增技术与Smart-seq2的扩增技术有什么区别?
答: Smart-seq2扩增技术针对是的3’~5’端全部的mRNA;3’端扩增技术主要是扩增3’端,是否能延伸至5’端,取决于酶的活性等系列实验因素决定。因此,扩增产物的长度上会有一定的区别和差异。
Q5. 细胞物种没有相应的参考基因组,可以做这个产品吗?
答: 产品分析需要基于一个良好注释和组装的参考序列。基因注释不完善或只注释到转录本,没有注释到基因等的不完善参考序列,会出现占用内存过多,即使对转录本进行去冗余及重新构建,跑出的结果可能会不太好,需要客户明确其风险因素。
Q6. DNBelab C4平台产生的Raw data是采用什么平台分析?
答:DNBelab C4有配套的数据分析软件,可以提供给客户,另外,可以注册华大DCS智能云平台,在云平台上进行数据分析。