SeqArc
SeqArc User Manual
Chapter 1: Product Information
1.1 Product Description
SeqArc is a high-efficiency compression tool specifically designed for genomic sequencing data, offering exceptional compression performance for FASTQ format files. It utilizes reference genome information for sequence alignment or employs statistical models for reference-free compression, balancing compression ratio and performance.
Through multi-threaded parallel processing, SeqArc significantly reduces file size while ensuring data integrity. It supports lossless compression for all species, maintaining both high compression rates and fast compression/decompression speeds, effectively handling both short-read and long-read sequences.
Key Features:
Dual-mode compression: Supports reference genome-assisted compression and reference-free statistical compression.
Classification indexing: Utilizes multiple reference genomes to build classification indexes, improving compression rates.
High-performance processing: Multi-threaded parallel compression and decompression for enhanced speed.
Data security: Supports data integrity verification during compression and decompression.
Flexible output: Supports standard output or multiple file formats during decompression.
1.2 Notes
- Index construction: Building indexes for large reference genomes may require significant time and memory.
- Data verification: Enabling compression and decompression verification enhances data security but increases processing time.
- File overwriting: Without the -f/--force option, the program will not overwrite existing output files.
- Temporary files: Compressing multiple files creates temporary directories; ensure sufficient disk space is available.
- Reference selection: Choosing an appropriate reference genome can significantly improve compression rates.
Chapter 2: Product Introduction
SeqArc provides three core functions:
- Index construction: Builds indexes for reference genomes or classification databases.
- Data compression: Compresses FASTQ files into the proprietary .arc format.
- Data decompression: Restores .arc files back to FASTQ format.
2.1 Index Construction
Constructing indexes is a critical preliminary step to improve compression rates. Prepare the reference sequences for the species corresponding to the FASTQ files, then execute the construction process.
2.2 Compression Flowchart
SeqArc independently compresses the three parts of FASTQ files—IDs, sequences, and quality values. For sequence compression, in addition to entropy encoding, it provides a sequence alignment module based on HASH or minimimap2 to enhance the compression rate for successfully aligned sequences. The design of data blocks balances compression rates and performance, while the introduction of encapsulation formats ensures data security, independent access to partial data, and backward compatibility.
Below is the compression flowchart:

2.3 Decompression Flowchart
SeqArc reads each compressed block based on the data compression information recorded in the compressed file, sequentially restoring IDs, sequences, and quality values. These are then combined into complete FASTQ data blocks, followed by checksum calculations and comparison with the original recorded checksums to ensure decompression accuracy.
Below is the decompression flowchart:

Chapter 3: User Guide
SeqArc fully manages compression and decompression processes through the DCS Intelligent Cloud Platform. Below is the operational guide for using SeqArc on the DCS Intelligent Cloud Platform.
3.1Guide Overview
3.1.1 Overview
This chapter explains how to use SeqArc for FASTQ data compression and decompression. Before use, carefully read and understand the content to ensure correct usage.
3.1.2 Process Suite
- seqarc_index
- seqarc_compress
- seqarc_decompress
3.2 Usage Scenarios
The operation consists of five steps: uploading data, adding workflows, constructing indexes, executing compression workflows, and executing decompression workflows. When the task status shows "completed," the task is finished.
3.2.1 Uploading Data
- Click the left navigation bar [Data Management], enter the data management page, navigate to the target folder, and click the upper-right corner [+ Add File] - [Tool Upload] to upload data (Figure 3-1):
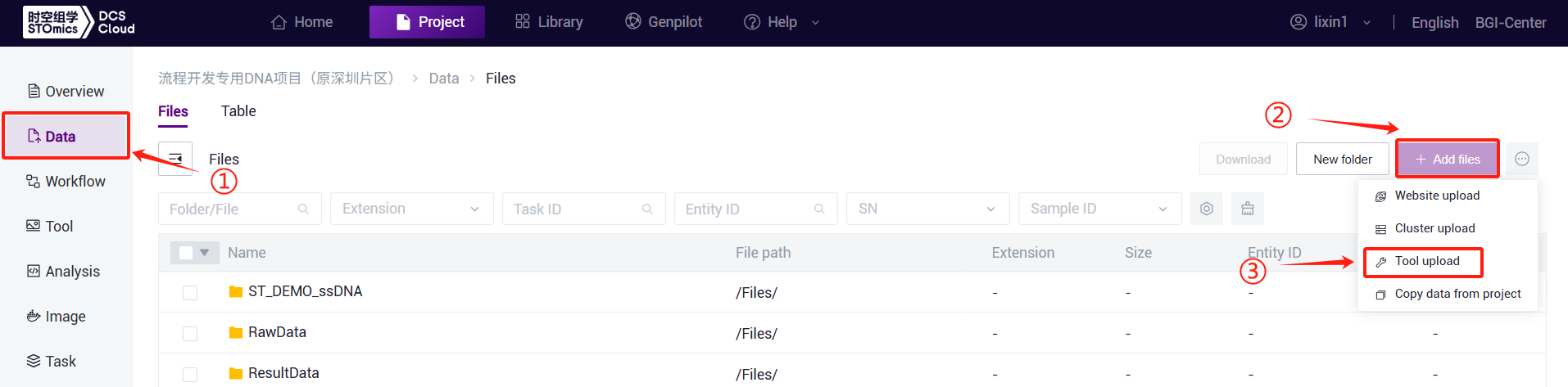
- Click [Upload], browse and select the required files. After uploading, the files will appear in the target folder (Detailed guidance is available here: https://cloud.stomics.tech/helpcenter/usermanual/data.html#add-files):
3.2.2 Adding Workflows
- Click the top navigation bar [Project] to enter the project list page. Enter the project name/project number in the search box and click to enter the project (Figure 3-2):
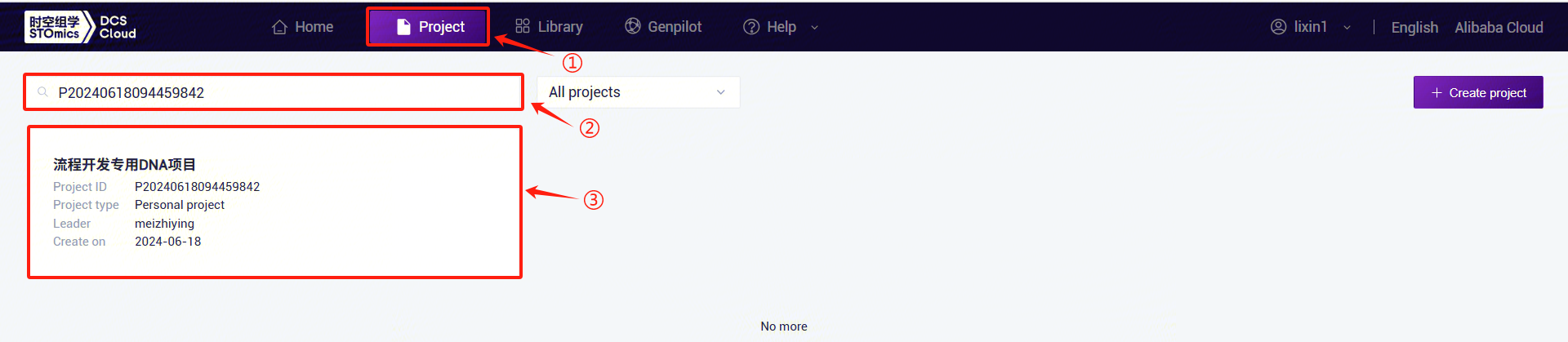
- Click the left navigation bar [Process Analysis] to enter the process analysis page. Click the upper-right corner [+ Add Workflow] to add a workflow, then click [Copy from Public Library] (Figure 3-3):
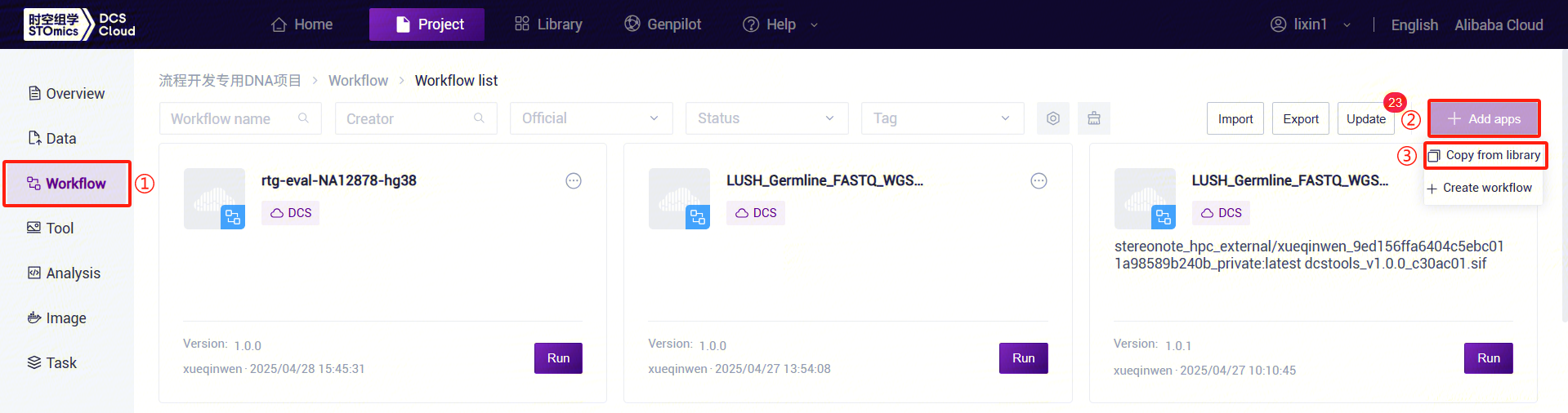
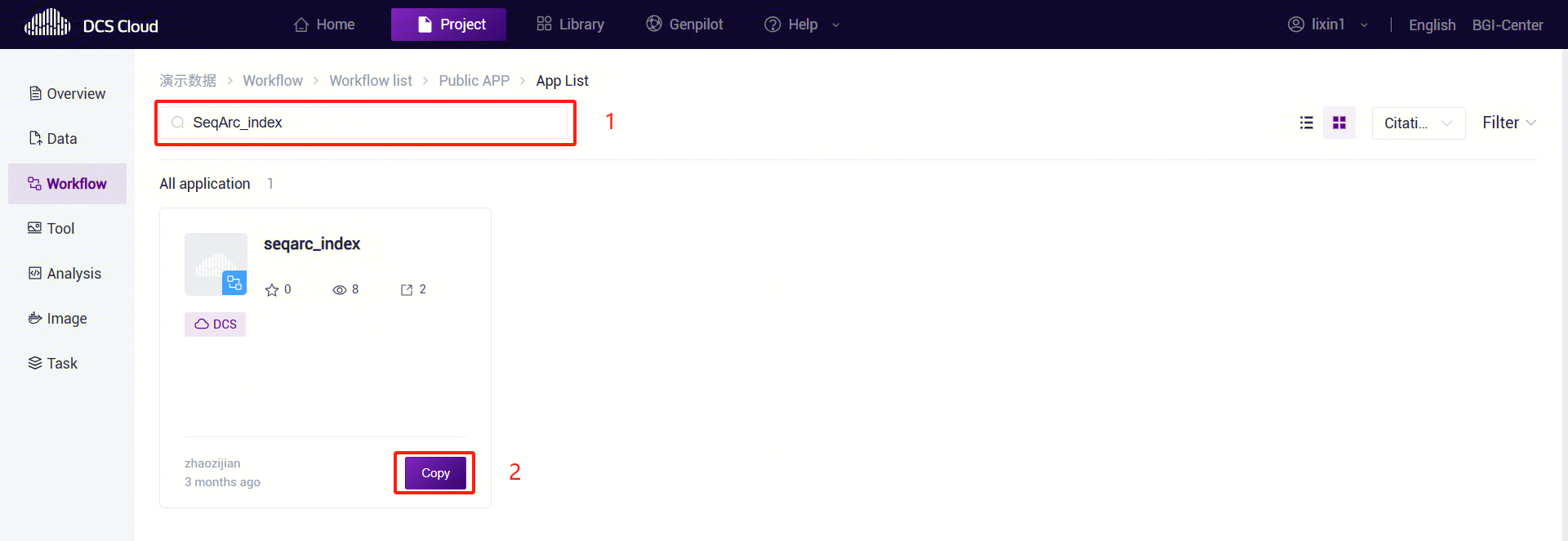
- Usingseqarc_index as an example, enter the workflow name 'seqarc_index' in the search box, select it, click [Copy], then click [OK]. Repeat the same steps for other process suites:
3.2.3 Constructing Compression Reference Index with seqarc_index
- Click the left navigation bar**[Process Analysis]**, enter the process analysis page, search for seqarc_index, and click [Run]:

- Enter the entity ID and click**[Next]** (Figure 3-6):

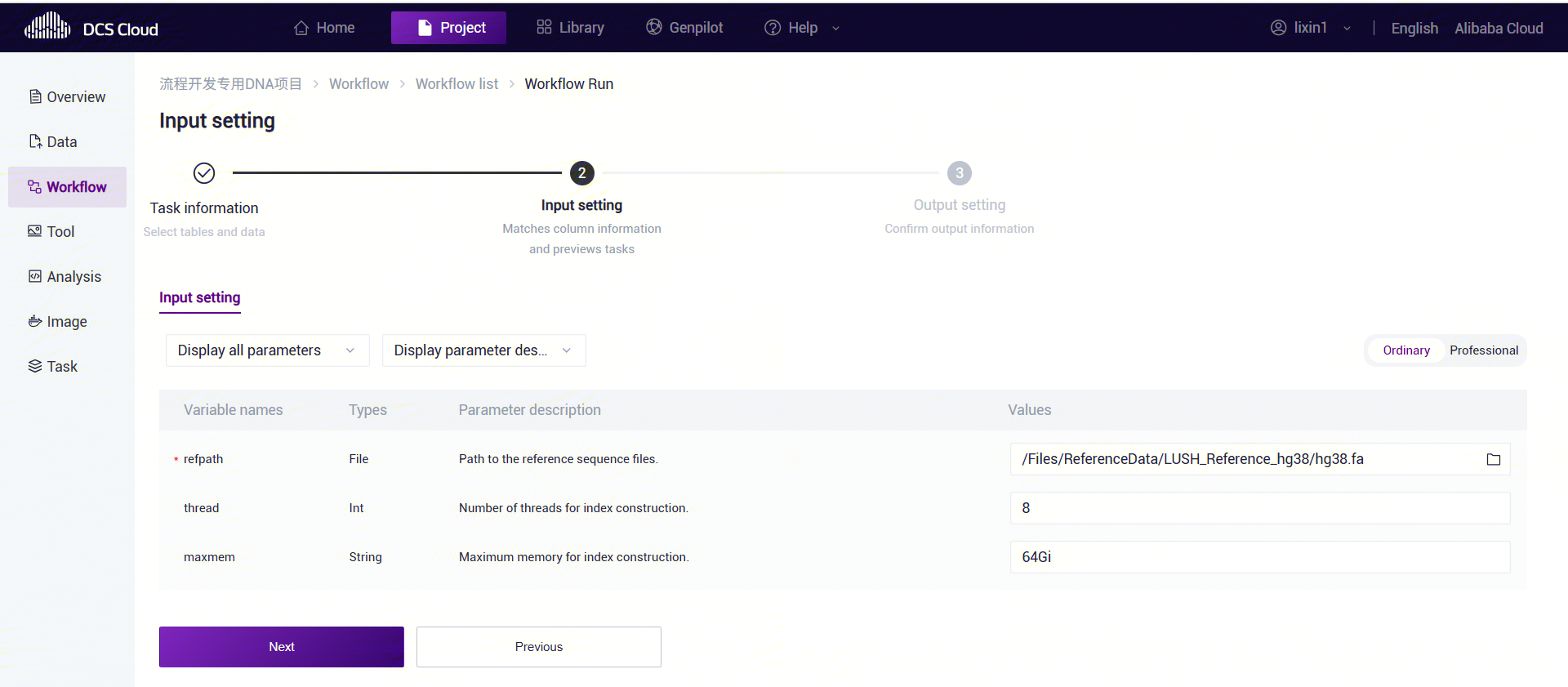
seqarc_index** **Variable Descriptions:
- refpath: Path to the reference sequence files.
- thread: Number of threads for index construction.
- maxmem: Maximum memory for index construction.
- Click [Run] to start the analysis (Figure 3-8):
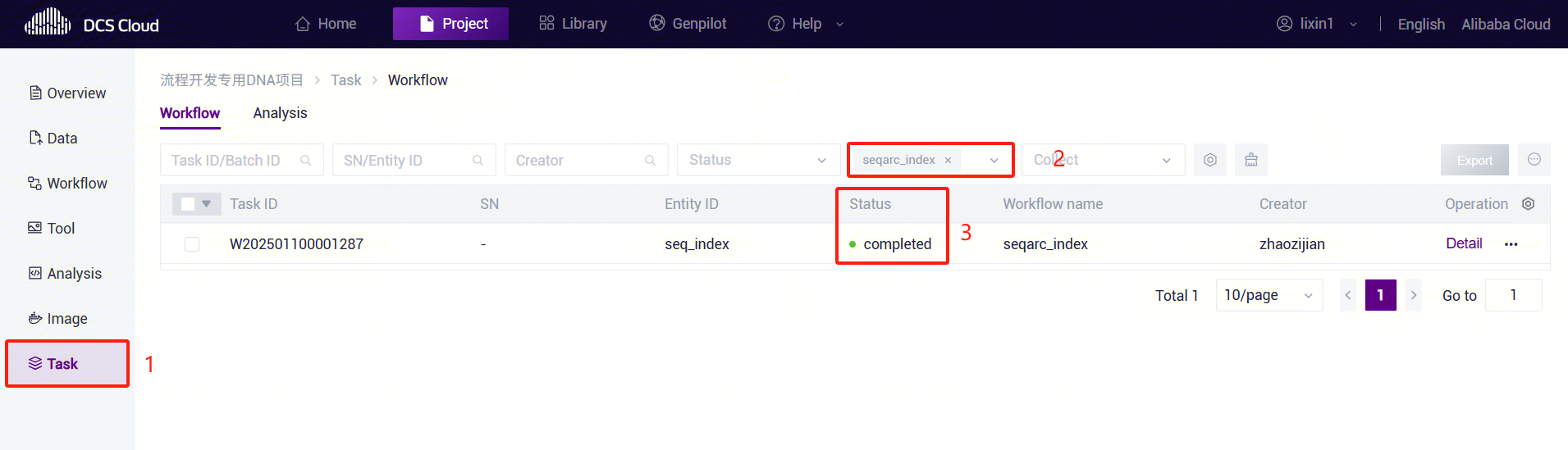
- Click the left navigation bar [Task Management] to enter the process analysis page. Select seqarc_index under Process Name to view the task status. Once completed, the task status will display as "completed." Copy the task number, click the navigation bar [Data Management], and enter the task number in the folder/filename search box. Navigate to the result files, where the reference folder serves as the input for the refpath parameter in the seqarc_compress and seqarc_decompress workflows.
3.2.4 Constructing Index Step 5
- Click the left navigation bar [Process Analysis], enter the process analysis page, search for seqarc_compress, and click [Run] (refer to 3.2.3).
- Enter the entity ID (usually the sample ID) and click [Next] (refer to 3.2.3):

- Enter the FASTQ path, reference sequence path, and name, then click [Next]:

seqarc_compress Parameter Descriptions:
- inputfq: Path to the input FASTQ file.
- outname: Output file name (the workflow automatically appends the .arc suffix).
- refpath: Path to the reference sequence files.
- thread: Number of threads for compression.
- maxmem: Maximum memory for compression.
3.2.5 ARC Decompression Workflow
- Click the left navigation bar [Process Analysis], enter the process analysis page, search for seqarc_decompress, and click Run] (refer to 3.2.3).
- Enter the entity ID (usually the sample ID) and click [Next] (refer to 3.2.3):

- Enter the ARC file to decompress and the corresponding reference sequence, then click [Next]:

seqarc_decompress** **Parameter Descriptions:
- inputarc: Path to the input ARC file.
- outname: Output file name.
- refpath: Path to the reference sequence files.
- thread: Number of threads for decompression.
- maxmem: Maximum memory for decompression.